Dégradation des protéines
Dégrons internes et autres signaux d’ubiquitination
Phospho-dégrons et dégrons conformationnels
- Biochimie
- Chimie organique
- Bioénergétique
- Composition de la matière vivante
- Composés organiques
- Protides
- Acides nucléiques
- Glucides
- Lipides
- Hormones
- Composés inorganiques
- Composés organiques
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

Les dégrons internes et autres signaux d’ubiquitination orchestrent la reconnaissance sélective et la dégradation des protéines par le protéasome, assurant une régulation fine de leur stabilité et de leur fonction.
Vue d'ensemble
Les dégrons internes et autres signaux d’ubiquitination constituent une classe essentielle de déterminants de la stabilité protéique, distincte des signaux terminaux.
1. Contrairement aux N-dégrons et C-dégrons, ce sont des motifs courts ou des régions structurales localisés à l’intérieur de la séquence polypeptidique qui conditionnent l’ubiquitination en fonction de signaux intracellulaires variés comme :
- la phosphorylation,
- les changements conformationnels
- l’interaction avec des partenaires spécifiques.
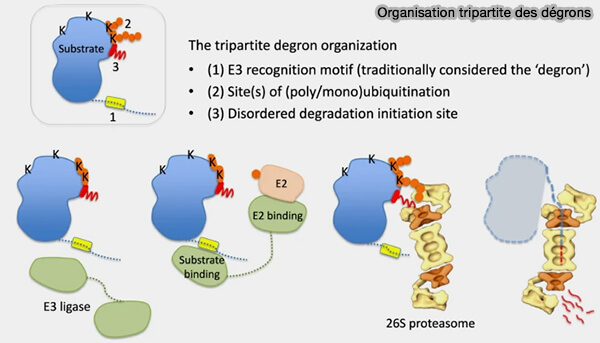
(Figure : vetopsy.fr d'après Guharoy et coll)
2. Cette diversité permet à la cellule de réguler de manière fine et dynamique la dégradation de protéines clés impliquées dans :
- le cycle cellulaire et la prolifération,
- la signalisation,
- la différenciation,
- le contrôle qualité.

Les dégrons internes du cycle cellulaire, étudiés dans un chapitre spécifique, agissent comme des interrupteurs moléculaires, coordonnant la destruction sélective des régulateurs du cycle cellulaire.
Phospho-dégrons
Les phospho-dégrons représentent une classe de signaux de dégradation dont la reconnaissance dépend directement d’une modification post-traductionnelle, en général une phosphorylation d’un ou de plusieurs groupements phosphate sur des résidus sérine, thréonine ou tyrosine (Phosphodegrons in Health and Disease: From Cellular Homeostasis to Therapeutic Potential 2025).
Contrairement aux dégrons constitutifs, les phospho-dégrons sont activés uniquement après l’ajout d’un ou de plusieurs groupements phosphate sur des résidus sérine, thréonine ou tyrosine (
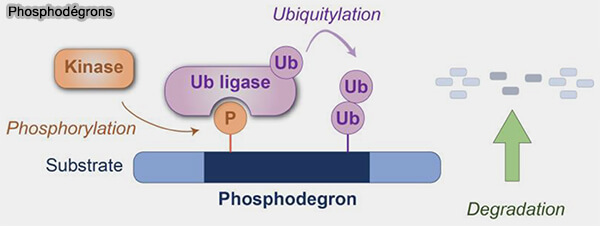
(Figure : vetopsy.fr d'après Nakagawa et coll)
1. Ils traduisent l’activité kinase-phosphatase de la cellule en signaux protéolytiques pour la coupler à :
- à des voies de signalisation spécifiques, extracellulaires ou intracellulaires, à la protéolyse,
- à l’état d’activation de certaines kinases,
- à la progression du cycle cellulaire et à la régulation de la transition entre les phases du cycle,
- à l’activation de réponses au stress,
- au contrôle de la durée de vie de protéines à activité transitoire.
2. Ils confèrent ainsi un niveau supplémentaire de contrôle spatio-temporel, garantissant que la dégradation n’a lieu que dans des contextes physiologiques précis.
CPD (Cdc4 phospho-dégron)
Le CPD est un dégron phosphorylé reconnu par la ligase SCFCdc4, composé d’un consensus (S/T)PXX(S/T), dans lequel le résidu sérine ou thréonine phosphorylé est essentiel à la liaison de l’adaptateur Cdc4 via son domaine WD40 (Cdc4 phospho-degrons allow differential regulation of Ame1CENP-U protein stability across the cell cycle 2021).
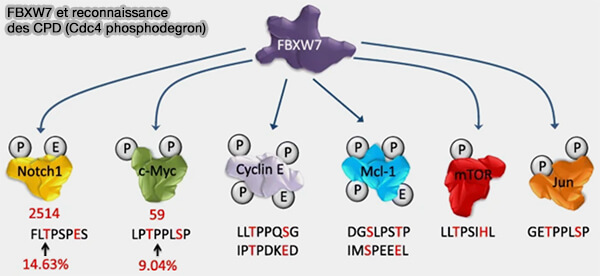
(Figure : vetopsy.fr d'après Yeh et coll)
Le CDP intervient dans la dégradation d’inhibiteurs de CDK (CK1) tels que p27Kip1, dont l’élimination est nécessaire à la progression de la phase G1 vers la phase S.
- Au cours de la phase G1 tardive, p27Kip1 est phosphorylé sur la thréonine 187 par le complexe cycline E/CDK2, créant le motif phosphorylé reconnu par le complexe SCFSkp2, ou SCFCdc4 selon les espèces.
- Cette interaction conduit à sa polybiquitination K48 et à sa dégradation par le protéasome 26S, levant ainsi l’inhibition exercée sur les complexes cycline E/CDK2 et cycline A/CDK2, nécessaires à l’entrée en phase S et à la réplication de l’ADN.
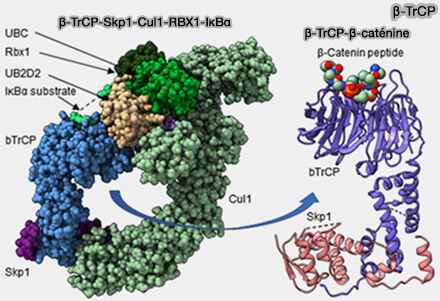
β-TrCP dégron (DSGxxS)
Le dégron β-TrCP est un motif doublement phosphorylé reconnu par la ligase SCFβ-TrCP, caractérisé par la séquence consensus DSGxxS (A systems-wide screen identifies substrates of the SCFbTrCP ubiquitin ligase 2013).
Il joue un rôle central dans la régulation.
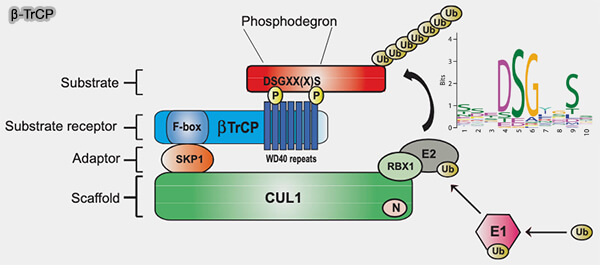
(Figure : vetopsy.fr d'après Low et coll)
1. Dans la voie NF-κB, la phosphorylation d’IκBα induit sa reconnaissance par SCFβ-TrCP, entraînant son ubiquitination et sa dégradation protéasomale.
(Figure : vetopsy.fr d'après Jaffry et Wells)
Cette destruction libère le facteur NF-κB, qui peut alors migrer vers le noyau et activer la transcription de gènes cibles.
2. Dans la voie Wnt/β-caténine, en conditions basales, β-TrCP reconnaît le motif phosphorylé de la β-caténine, résultant de l’action séquentielle des kinases CK1 et GSK3β (Small molecule and peptide inhibitors of βTrCP and the βTrCP–NRF2 protein–protein interaction 2023).
Cette interaction conduit à la dégradation de β-caténine par le protéasome, empêchant l’activation transcriptionnelle des gènes Wnt-dépendants.
Sites GSK3β
1. Certains phospho-dégrons nécessitent une phosphorylation séquentielle dite hiérarchique.
a. Une première kinase, le plus souvent une CDK ou CK1, phosphoryle un résidu d’amorçage, qui crée un site de recrutement et d’activation pour GSK3β.
b. GSK3β réalise la phosphorylation secondaire indispensable à la reconnaissance par la ligase E3.
GSK3β ne peut phosphoryler son substrat que si un résidu en position +4 est déjà phosphorylé.
c. La double phosphorylation forme alors le phospho-dégron complet de haute spécificité pour les ligases E3, le plus souvent de type SCF (β-TrCP, Fbxw7/Cdc4).
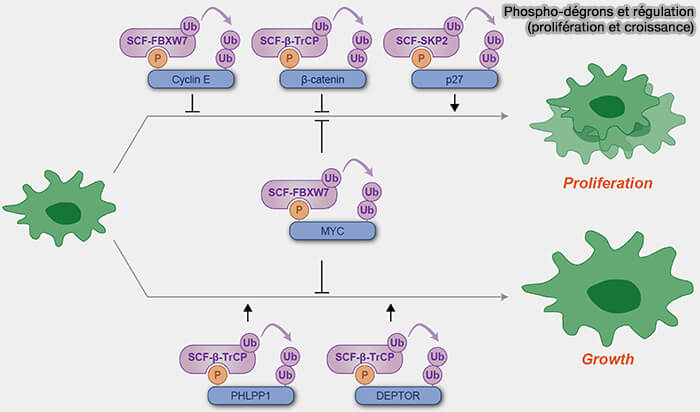
(Figure : vetopsy.fr d'après Nakagawa et coll)
2. Ce mécanisme contrôle la stabilité de nombreux substrats clés, notamment :
- les facteurs de croissance, i.e. IRS1, GS (glycogène synthase), TSC2,
- les régulateurs transcriptionnels, i.e. c-Myc, cycline E, c-Jun,
- les protéines du cycle cellulaire, i.e. cyclines D1/E, Cdc25A, claspine, impliquées dans la transition G1/S.
Dégrons conformationnels et structurels
Les dégrons conformationnels et structurels représentent une catégorie particulière de signaux de dégradation qui ne dépendent pas d’une séquence linéaire canonique, mais plutôt de la conformation tridimensionnelle de la protéine ou de l’état structural local de certaines de ses régions.
1. Ces dégrons traduisent l’état de repliement, l’intégrité structurelle ou la dynamique conformationnelle d’une protéine en un signal de dégradation.
Ces dégrons :
- assurent l’élimination des protéines mal conformées,
- préviennent l’accumulation de formes agrégées,
- participent au maintien de la protéostasie cellulaire.
2. Dans certains cas, leur exposition est régulée de manière réversible, ce qui permet d’intégrer des signaux conformationnels dans des programmes cellulaires tels que la différenciation ou la réponse au stress.
Dégrons dépendants du repliement
Les dégrons dépendants du repliement sont des signaux de dégradation générés lorsque la conformation native d’une protéine n’est plus maintenue.
L’exposition anormale de régions hydrophobes internes, normalement enfouies dans le cœur de la structure tridimensionnelle, constitue le marqueur central indiquant un mauvais repliement ou une instabilité structurelle.
1. L’exposition de régions hydrophobes internes, normalement enfouies dans le cœur de la protéine, constitue un puissant signal de dégradation lors de :
- mauvais repliements consécutifs à des erreurs de traduction ou à un stress cellulaire (
repliement des protéines),
- mutations déstabilisantes altérant la structure tertiaire,
- agrégations partielles ou exposition transitoire d’éléments hydrophobes après dénaturation.
Ces signaux apparaissent très en amont dans la surveillance qualité et constituent la première alerte reconnaissable par la cellule.
2. Ces régions hydrophobes exposées sont détectées par des ubiquitine-ligases spécialisées, souvent couplées à des chaperonnes moléculaires, capables de distinguer les conformations natives des conformations aberrantes.
- Dans le cytosol, des complexes comme CHIP (C-terminus of Hsp70-interacting protein), en association avec Hsp70 ou Hsp90, assurent la sélection et l’ubiquitination des protéines instables.
- Dans le réticulum endoplasmique, le système ERAD (ER-Associated Degradation), faisant intervenir des ligases comme HRD1, gp78 ou Doa10, cible et élimine les protéines mal repliées exportées vers le cytoplasme pour leur dégradation par le protéasome.
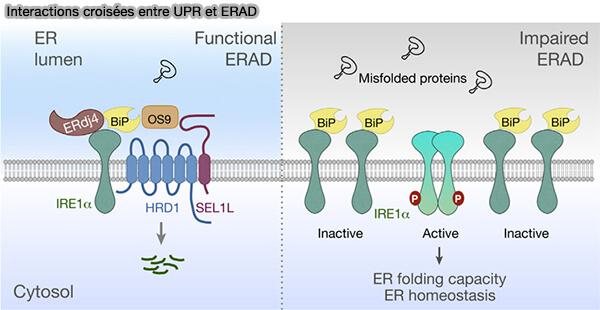
(Figure : vetopsy.fr d'après Hwang et Qi)
3. Les dégrons de repliement constituent la première alerte signalant la présence d’intermédiaires non conformes.
a. Ils orientent prioritairement les substrats vers les mécanismes locaux de contrôle qualité, en particulier l’ERAD pour les protéines luminales ou membranaires du RE (
b. Lorsque la quantité de protéines portant ces dégrons dépasse les capacités d’élimination du RE, la cellule active la réponse UPR (
Contrairement à l’ERAD, centrée sur des substrats individuels, l'UPR constitue une réponse adaptative globale, visant à augmenter la production de chaperonnes, à renforcer l’efficacité de l’ERAD et à réduire temporairement l’afflux de protéines dans le RE.
c. Ainsi, les dégrons de repliement jouent le rôle de signal initial, l’ERAD celui de mécanisme d’élimination local, et la UPR celui de mécanisme systémique maintenant la protéostasie lorsque la charge devient excessive.
4. En détectant et éliminant les protéines dénaturées, mal repliées ou agrégées, ces mécanismes :
- préservent l’intégrité des complexes multiprotéiques,
- empêchent la formation d’agrégats cytotoxiques,
- évitent la saturation des systèmes de dégradation,
- préviennent l’installation d’un stress chronique du RE.
PEST
Le motif PEST est une séquence riche en proline (P), acide glutamique (E), sérine (S) et thréonine (T), souvent localisée dans des régions désordonnées des protéines.
Il agit comme un signal de déstabilisation favorisant la dégradation rapide des protéines à demi-vie courte.

Le motif PEST est étudié dans un chapitre spécifique.
1. Sa phosphorylation peut transformer cette région en phospho-dégron, reconnu par certaines ligases E3 de type SCF, reliant ainsi sa fonction à la signalisation cellulaire.
2. Certains domaines PEST jouent également un rôle important dans la régulation du cycle cellulaire (
Motif PY (PPxY)
1. Le motif PY, défini par la séquence consensus PPxY, i.e. proline (P) et tyrosine (Y), est localisé dans des régions flexibles ou désordonnées des protéines et agit comme un site d’interaction spécifique pour certaines E3 ubiquitine ligases contenant un domaine WW.
- Le motif PY présente une géométrie locale en polyproline II, principalement portée par la di-proline (PP), qui rigidifie le segment et optimise sa reconnaissance par les domaines WW des ligases E3 de la famille NEDD4.
- Sa tyrosine terminale est essentielle pour l’ancrage du motif : sa mutation ou sa phosphorylation peut moduler la liaison avec la ligase.
1. Sa plasticité et sa présence dans des contextes fonctionnels variés en font un module d’adressage et de régulation plus qu’un dégron classique au sens strict.
a. Le motif PY joue un rôle clé dans la connectivité entre les voies de signalisation et la régulation de la protéostasie, en traduisant les interactions protéine-protéine en signaux d’ubiquitination qui peuvent conduire selon la nature du substrat et le contexte cellulaire :
- soit à la dégradation protéasomale,
- soit à la régulation du trafic membranaire.
b. Les ligases E3 de la famille NEDD4 (Notch, Smurf1/2, ITCH, WWP1/2), ligases HECT, reconnaissent le motif PY via leur domaine WW et catalysent l’ubiquitination du substrat.
- Dans la voie Notch, le motif PY intervient dans la régulation de l’endocytose et du recyclage du récepteur.
- Dans la signalisation TGF-β, les ligases Smurf1 et Smurf2 reconnaissent des substrats PY tels que Smad7, favorisant leur ubiquitination et leur élimination.
- Dans la voie Hippo, les protéines d’adhérence, comme LATS1 et LATS2 contiennent également des motifs PY régulant leurs interactions avec les ligases WWP1 ou ITCH.
Remarque : le motif PY, en reliant les voies de signalisation Notch, TGF-β et Hippo à la machinerie ubiquitine-protéasome, intervient également dans la régulation de :
- l’endocytose,
- la dynamique du cytosquelette.


