Cycle cellulaire
Reproduction cellulaire
Crossing-over
- Biologie cellulaire et moléculaire
- Constituants de la cellule
- Reproduction cellulaire
- Biochimie
- Transport membranaire
- Moteurs moléculaires

Le crossing-over est un échange réciproque d’ADN entre chromatides non sœurs de chromosomes homologues en méiose I, indispensable à la diversité génétique et à la ségrégation correcte.
Le crossing-over assure deux fonctions majeures :
- la diversité génétique des gamètes,
- la stabilité mécanique de la division réductionnelle (
méiose I).
Il constitue ainsi un point de convergence entre architecture chromosomique, recombinaison de l’ADN et fidélité de transmission génétique.
Vue d'ensemble
Un crossing-over correspond à un échange physique d’ADN entre deux chromatides non soeurs, i.e. appartenant à des chromosomes homologues.
Il résulte d’événements de recombinaison établis préférentiellement entre homologues, dont le choix est conditionné par l’architecture méiotique (
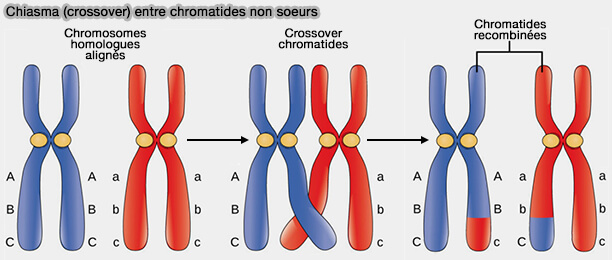
(Figure : vetopsy.fr d'après courses.lumenlearning.com)
1. En méiose I, contrairement à la mitose, ce n’est pas une simple réparation de cassure.
Le crossing-over constitue un événement structurant qui s’engage dès le début de la prophase I, initié au leptotène, poursuivi au zygotène, puis sélectionné et maturé au pachytène et qui produit :
- un échange de matériel génétique entre chromatides non sœurs,
- un lien physique et mécanique entre homologues,
- la formation d’un chiasma en diplotène, indispensable à l’alignement et à la séparation correcte des homologues.
2. Les chiasmas assurent la cohésion fonctionnelle entre les paires de chromosomes homologues appariés jusqu’à la métaphase I et conditionnent l’orientation correcte des chromosomes sur le fuseau.
En l’absence de crossing-over :
- les homologues ne restent pas attachés,
- leur orientation devient aléatoire,
- le risque de non-disjonction augmente fortement,
- la méiose I devient instable.
3. Le crossing-over est une nécessité mécanique pour la division réductionnelle.
Le chiasma forme un lien mécanique permettant la transmission des forces du fuseau entre les deux homologues et assure leur bi-orientation correcte en métaphase I.
Appariement des chromosomes homologues
L’appariement des chromosomes homologues constitue une étape indispensable à l’établissement des événements de recombinaison conduisant aux crossing-over et repose sur une reconnaissance homologue initiée aux stades leptotène-zygotène.
1. La reconnaissance entre chromosomes homologues en prophase I repose sur trois mécanismes complémentaires.
- La mise en contact des chromosomes homologues dans le noyau repose sur la dynamique spatiale des chromosomes, liée au bouquet télomérique et aux mouvements dépendants du cytosquelette nucléaire, via le complexe LINC, qui rend la reconnaissance homologue efficace.
- Les cassures double brin programmées génèrent des extrémités d’ADN simple brin servant de support moléculaire à la recherche d’homologie et à l’invasion de brin.
- Le choix préférentiel du chromosome homologue repose sur l’action des recombinases RAD51 et DMC1, qui utilisent les extrémités d’ADN simple brin pour tester la complémentarité de séquence avec l’ADN du partenaire homologue et favoriser les interactions inter-homologues plutôt qu’inter-sœurs.
2. Ces processus agissent de concert pour rendre la reconnaissance efficace, avant l’installation complète du complexe synaptonémal, qui intervient ensuite comme élément structural de stabilisation et de maturation de l’appariement.
Mise en contact des homologues dans le noyau
La reconnaissance entre chromosomes homologues dépend non seulement des mécanismes moléculaires de recombinaison, mais aussi de la dynamique spatiale des chromosomes dans le noyau.
L’organisation nucléaire précoce contribue ainsi activement à l’entrée en prophase I et à la mise en place des conditions favorables à l’appariement.
1. Avant et au tout début de la prophase I, l’organisation spatiale des chromosomes dans le noyau crée un contexte favorable à leur rapprochement, préparant la mise en place ultérieure du bouquet télomérique.
Remarque : l’enveloppe nucléaire restant intacte durant toute la prophase I, ces interactions télomériques peuvent persister jusqu’au diplotène, avant de disparaître lors de la rupture de l’enveloppe (NEBD, Nuclear Envelope Breakdown) à l’entrée en métaphase I.
2. Lors de l'entrée en prophase I, l'organisation spatiale initiale est ensuite remodelée lors de l’entrée en prophase I, conduisant à l’établissement fonctionnel du bouquet télomérique, i.e. attachement transitoire des télomères à l’enveloppe nucléaire, s’organisant en une structure polarisée appelée bouquet télomérique.

Vous pouvez lire : Centromere positioning orchestrates telomere bouquet formation and the initiation of meiotic differentiation (2025), article dans lequel les mécanismes sont détaillés.
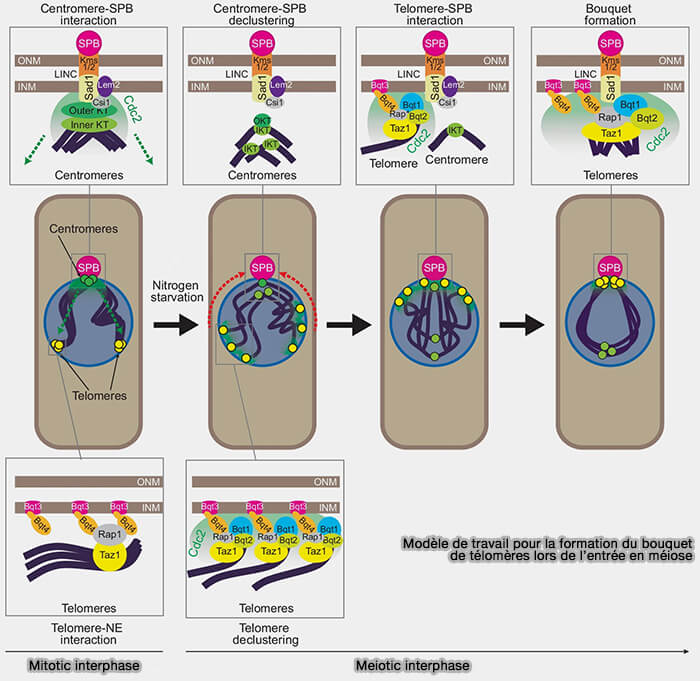
(Figure : vetopsy.fr d'après Jiménez-Martín et coll)
Sur la figure, chez la levure Schizosaccharomyces pombe, les chromosomes adoptent en interphase une configuration de type Rabl, caractérisée par un regroupement des centromères à proximité du corps du pôle du fuseau (SPB) via les complexes kinétochore-LINC, tandis que les télomères occupent une position opposée dans le noyau. Cette organisation n’est pas neutre : elle constitue un état spatial préconfiguré qui oriente la dynamique nucléaire lors de l’entrée en méiose.
a. Lors de l’engagement méiotique, une dissociation partielle du kinétochore externe se produit, entraînant la libération des centromères de leur ancrage aux pôles du fuseau.
Cette libération s’accompagne d’une réorganisation de l’activité nucléaire et favorise la mobilisation progressive des télomères le long de l’enveloppe nucléaire.
b. Les télomères sont alors recrutés aux complexes LINC (Linker of Nucleoskeleton and Cytoskeleton) via des protéines spécifiques du bouquet, telles que Bqt1 et Bqt2 chez la levure.
- Ce couplage relie physiquement les chromosomes au cytosquelette, ce qui induit des mouvements actifs du noyau et des chromosomes.
- Ces mouvements conduisent progressivement au regroupement polarisé des télomères sous le SPB, formant le bouquet télomérique caractéristique du leptotène-zygotène.
3. Cette configuration polarisée a plusieurs conséquences fonctionnelles majeures :
- elle augmente la probabilité de collision entre chromosomes homologues,
- elle réduit l’espace de recherche tridimensionnel,
- elle favorise la stabilisation des premières interactions homologues,
- elle facilite l’installation ultérieure du complexe synaptonémal.

Les mutants dépourvus de bouquet télomérique ou de mouvements nucléaires présentent une synapsis retardée, incomplète ou incorrecte.
Reconnaissance homologue et engagement de la recombinaison
Cassures double brin (DSB) programmées :
rôle mécanique dans la reconnaissance
Les chromosomes méiotiques s’organisent en axes protéiques longitudinaux entourés de boucles de chromatine.
- Lorsque la cellule s’engage en prophase I, un ensemble de gènes strictement spécifiques de la méiose est exprimé.
- Ce programme méiotique est coordonné avec la formation des axes chromosomiques, la réorganisation de la chromatine et la mise en place de la recombinaison.
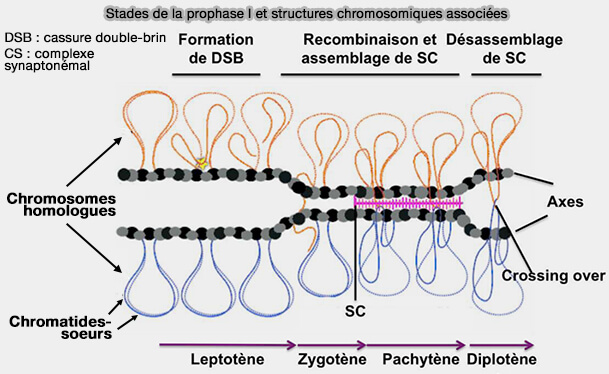
(Figure modifiée : vetopsy.fr d'après Yadav et Bouuaert)
1. PRDM9 est exprimée spécifiquement au leptotène-zygotène de la prophase I, puis disparaît (PRDM9, a driver of the genetic map 2018).
a. Elle se fixe alors transitoirement sur des motifs de séquence précis situés dans les boucles de chromatine, grâce à son domaine à doigts de zinc (Caractérisation biochimique des protéines REC114, MEI4 et IHO1 impliquées dans l’initiation de la recombinaison méiotique chez la souris 2022).
Quand PRDM9 se fixe à son motif, elle méthyle les histones H3K4 et H3K36 (H3K4me3, H3K36me3) sur les nucléosomes voisins.
b. Les protéines promotrices de la formation des DSB, telles que HORMAD1, MEI4, REC114 et IHO1, situées sur l'axe chromosomique et un facteur intermédiaire, qui pourrait être CxxC1, assureraient l'interaction de PRDM9 avec la machinerie de formation des DSB sur l’axe chromosomique.
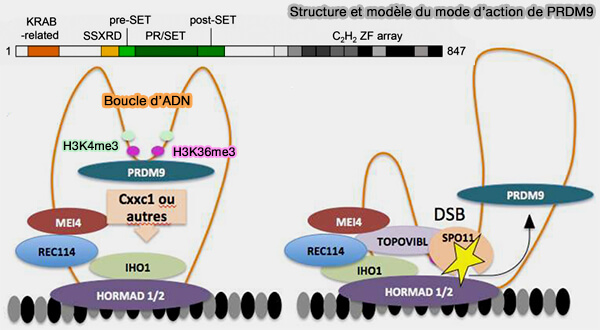
(Figure modifiée : vetopsy.fr d'après Grey et coll)
2. Les marques déposées par PRDM9 transforment localement l’environnement chromatinien et définissent les futurs hotspots de recombinaison conditionnant le recrutement de la machinerie de formation des DSB, incluant Spo11.
- Spo11 agit comme la sous-unité catalytique d’une topoisomérase de type VI, en association avec TOPOVIBL (également appelée MTOPVIB), formant le complexe enzymatique responsable des cassures double brin méiotiques (Mechanism and Control of Meiotic DNA Double-Strand Break Formation in S. cerevisiae 2021).
- Les protéines promotrices de la formation des DSB assurent le recrutement et le positionnement spatial de Spo11 aux sites où les cassures doivent être introduites.
Sans ces partenaires, Spo11 est présente mais catalytiquement inactive.
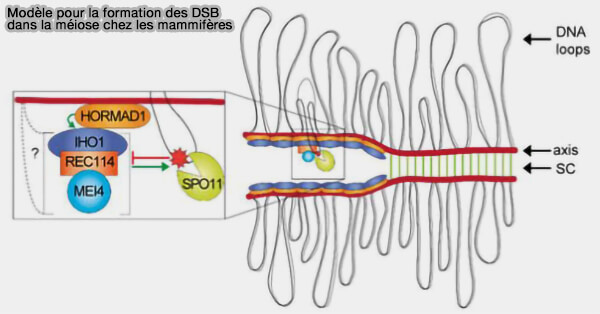
(Figure modifiée : vetopsy.fr d'après Stanzione et coll)
3. Une fois activée et recrutée, Spo11 introduit une cassure double brin (DSB) sur l’ADN d’une chromatide au sein d’un chromosome (Meiotic DNA break formation requires the unsynapsed chromosome axis-binding protein IHO1 (CCDC36) in mice 2016).
a. Cette cassure est ensuite maturée par des nucléases qui résectent les extrémités et produisent des queues simple brin 3’.
b. Ces extrémités simple brin constituent le support moléculaire de la reconnaissance homologue.
Elles deviennent capables d’interagir de manière stable uniquement avec une région d’ADN présentant une forte identité de séquence, c’est-à-dire le chromosome homologue.
Sans cassure, il n’existe donc aucun support physique permettant la reconnaissance. Sans extrémité simple brin, il n’existe aucun mécanisme de test de complémentarité.
Choix du chromosome homologue lors de la recombinaison
Quand Spo11 induit une cassure sur une chromatide, la cellule a deux possibilités pour la réparer :
- utiliser la chromatide sœur, copie quasi parfaite située à proximité,
- utiliser le chromosome homologue.
Le complexe synaptonémal n’initie pas ce choix, mais il le consolide en stabilisant physiquement les interactions établies avec le chromosome homologue et en rendant durable l’appariement sur toute la longueur des chromosomes.

Le complexe synaptonémal est étudié dans un chapitre spécifique.
1. En méiose, la cellule doit privilégier l’homologue, car c’est ce choix qui permet la formation des crossing-over et assure la séparation correcte des chromosomes en méiose I.
a. Pour imposer ce choix, la cellule modifie l’organisation physique des chromosomes au cours de la prophase I.
Les chromatides sœurs deviennent moins accessibles, tandis que les chromosomes homologues sont rapprochés, alignés et maintenus en parallèle.
b. Cette organisation dépend notamment des protéines de l’axe chromosomique (cohésines méiotiques, ainsi que Hop1/Red1 chez la levure, HORMAD1/2 chez les mammifères), qui :
- limitent l’accès préférentiel à la chromatide sœur,
- favorisent la stabilisation des interactions avec l’homologue,
- modulent l’activité des recombinases RAD51 et DMC1 en faveur des échanges inter-homologues.
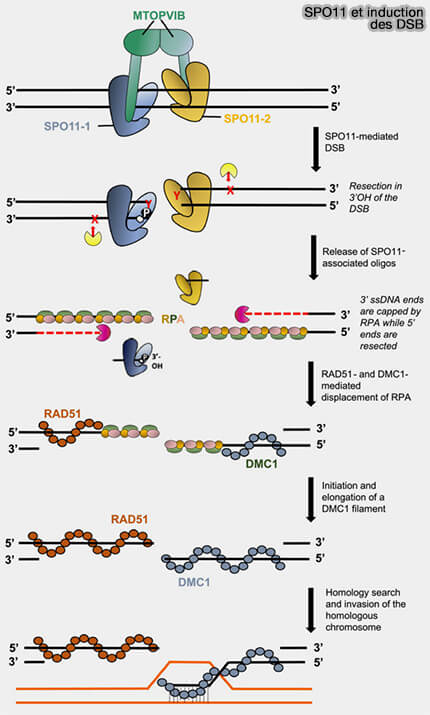
(Figure : vetopsy.fr d'après Fayos et coll)
2. Après la cassure induite par Spo11, les extrémités d’ADN simple brin sont prises en charge par deux protéines apparentées pour former un filament nucléoprotéique rigide (Engineering meiotic recombination pathways in rice 2019).
a. DMC1 est une recombinase spécifique de la méiose et est fortement activée en prophase I.
- DMC1 est favorisée par des cofacteurs méiotiques tels que HOP2-MND1, qui stabilisent son action et facilitent les échanges avec l’homologue.
- Elle possède une propriété essentielle car elle favorise les interactions avec le chromosome homologue, plutôt qu’avec la chromatide sœur.
b. RAD51 est la recombinase " classique ", utilisée aussi en mitose, mais ici son activité est partiellement freinée.
- À l’inverse, l’activité autonome de RAD51 est partiellement restreinte par des protéines associées aux axes chromosomiques, comme HORMAD1/2, ce qui limite la réparation préférentielle avec la chromatide sœur.
- Elle favorise naturellement la réparation en utilisant la chromatide sœur, car c’est la matrice la plus proche et la plus accessible.

Ce double contrôle oriente la recombinaison vers l’homologue et permet la formation des crossing-over.
3. Après le choix de l’homologue grâce à DMC1, une extrémité simple brin issue de la chromatide cassée :
- explore physiquement l’espace nucléaire,
- entre en contact transitoire avec d’autres doubles hélices,
- teste localement la complémentarité de séquence,
- déclenche l’invasion de brin uniquement si l’homologie est suffisante.
Le chromosome homologue est la matrice privilégiée car il possède une séquence suffisamment longue et parfaitement complémentaire pour stabiliser l’invasion de brin.
À ce stade, les deux chromosomes sont physiquement reliés par de l’ADN.
Organisation des crossing-over
Sélection et stabilisation des crossing-over
Une fois l’échange engagé, la cellule peut orienter la réparation vers deux types de produits :
- réparation sans échange de segments (non-crossing-over),
- réparation avec échange de segments (crossing-over)
En méiose, une partie des événements est volontairement orientée vers la formation de crossing-over, car ces échanges sont indispensables à la ségrégation correcte.
1. La cellule induit un excès de cassures au début de la prophase I par Spo11.
2. La cellule sélectionne une partie de ces événements pour être stabilisée en crossing-over, ce qui dépend de plusieurs facteurs.
a. Seules les régions situées dans des régions compatibles avec une contrainte mécanique efficace sont préférentiellement conservées.
- Les régions proches des centromères sont fortement défavorisées.
- Les régions très télomériques sont moins efficaces pour stabiliser un chiasma fonctionnel.
- Les zones intermédiaires, les bras chromosomiques, sont les plus propices.
b. Le complexe synaptonémal agit comme une plateforme de maturation des hotspots de recombinaison et seuls les événements qui s’intègrent le mieux dans sa structure ont le plus de chances d’être conduits vers un crossing-over.
c. Certaines protéines marquent les événements comme destinés à devenir des crossing-over en stabilisant certaines jonctions d’échange, telles que RNF212, HEI10 et le complexe MLH1-MLH3, qui :
- se concentrent sur un petit nombre de sites de recombinaison,
- stabilisent les intermédiaires moléculaires,
- empêchent leur résolution précoce en non-crossing-over.
Les autres cassures sont réparées sans échange de segments.
Interférence des crossing-over
Un mécanisme d’interférence empêche la formation de crossing-over trop proches les uns des autres, assurant leur répartition le long des chromosomes.
En amont de cette sélection, certains intermédiaires de recombinaison sont stabilisés par le complexe MutSγ, formé par MSH4 et MSH5, qui se fixe sur les jonctions de recombinaison et oriente ces événements vers la voie des crossing-over.

Ce contrôle garantit la présence d’au moins un chiasma par paire d’homologues, condition indispensable à la ségrégation correcte en méiose I.
1. Deux protéines, RNF212 et HEI10, jouent un rôle clé et agissent comme un système de marquage et de compétition entre sites de recombinaison selon plusieurs étapes (RNF212 is a dosage-sensitive regulator of crossing-over during mammalian meiosis 2014).
a. Au pachytène précoce, de nombreux sites de recombinaison sont engagés sur les chromosomes et sont tous potentiellement capables de devenir des crossing-over.
b. RNF212, ligase E3 de SUMO, se fixe transitoirement sur de nombreux sites de recombinaison le long du complexe synaptonémal et favorise la SUMOylation locale de facteurs, mais cette fixation est instable et compétitive.
- Certains sites recrutent un peu plus de RNF212 que d’autres.
- Ces sites deviennent progressivement plus stables.
c. HEI10, E3 ubiquitine ligase, agit ensuite comme un facteur d’amplification compétitive dont l'activité est modulée par HEIP1 (HEI10 interacting protein 1), qui facilite la dynamique des facteurs pro-crossing-over (HEIP1 orchestrates pro-crossover protein activity during mammalian meiosis 2025).
- Les sites qui recrutent davantage de RNF212 deviennent plus stables et correspondent à de futurs crossing-over.
- Les sites voisins perdent progressivement leurs facteurs et sont orientés vers une réparation sans crossing-over.
Cette compétition limite la proximité entre événements sélectionnés et assure une répartition régulière des crossing-over.
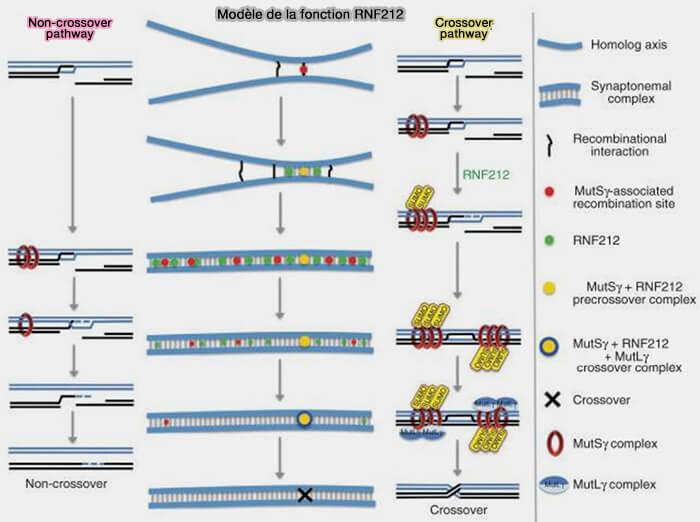
(Figure modifiée : vetopsy.fr d'après Stanzione et coll)
2. Les sites finalement sélectionnés par le couple RNF212/HEI10 recrutent ensuite le complexe MLH1-MLH3 (MutLγ), qui (Études de la régulation des interactions de la protéine MLH1 dans l’intégrité génétique des cellules méiotiques 2022) :
- stabilise définitivement l’intermédiaire,
- permet sa résolution finale en crossing-over.
MLH1 est d’ailleurs utilisé comme marqueur expérimental des sites de crossing-over en cytologie méiotique.
3. L’interférence des crossing-over repose sur une transmission physique du signal le long de la structure du complexe synaptonémal.
a. Le complexe ne constitue pas seulement un échafaudage passif : sa continuité structurale relie les sites de recombinaison d’un même bivalent et permet la redistribution de facteurs tels que RNF212 et HEI10 qui ne restent pas fixés sur un site, mais :
- s’associent aux sites de recombinaison,
- se détachent partiellement,
- diffusent sur une distance limitée le long du complexe synaptonémal, ce qui implique que l’inhibition est forte à proximité et s’atténue avec la distance,
- puis se réassocient ailleurs.
b. On parle de diffusion facilitée le long de la structure synaptonémale et cette organisation explique la sélection d’un site de crossing-over :
- limite mécaniquement la stabilisation de sites voisins
- contribue à l’espacement régulier des événements le long des chromosomes.
Conclusion
Le crossing-over apparaît ainsi comme un processus hautement coordonné, intégrant organisation chromosomique, dynamique nucléaire, recombinaison de l’ADN et architecture structurale.
Il ne résulte pas d’un événement isolé, mais d’une succession d’étapes contrôlées, depuis la reconnaissance des homologues jusqu’à la sélection des sites d’échange.
Cette intégration garantit à la fois la diversité génétique des gamètes et la fiabilité mécanique de la division méiotique, plaçant le crossing-over au cœur de la transmission héréditaire.
Retour vers la méiose
Biologie cellulaire et moléculaireConstituants de la celluleReproduction cellulaireMatériel génétiqueFuseauCycle cellulaireInterphasePhase G1Phase SPhase G2MitoseProphasePrométaphaseMétaphaseAnaphaseTélophaseCytokinèseAbcissionMéioseRégulation du cycle cellulaireCyclinesCDKComplexes Cyclines/CDKPoints de contrôles (checkponts)Enzymes mitotiquesMoteurs mitotiquesComplexes SMC (condensines et cohésines)BiochimieTransport membranaire Moteurs moléculairesVoies de signalisation


