Complexe APC/C
Structure : modules de reconnaissance et catalytique
- Biochimie
- Chimie organique
- Bioénergétique
- Composition de la matière vivante
- Composés organiques
- Protides
- Acides nucléiques
- Glucides
- Lipides
- Hormones
- Composés inorganiques
- Composés organiques
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

Le complexe APC/C assure sa spécificité et sa fonction enzymatique grâce à ses modules de reconnaissance et catalytique, qui orchestrent la sélection et l’ubiquitination sélective de ses substrats.
Les sous-unités APC/C sont organisées fonctionnellement et structurellement en quatre modules :
- le module central (APC1, 4, 5 et 15),
- le module TPR (APC3, 6, 7, 8 et APC12, 13, 16 et CDC26),
- le module de reconnaissance (APC10 et coactivateurs),
- le module catalytique (APC2, 11).
Le complexe APC/C fait partie des E3 ligases RING multiprotéiques " CRL-like ".
Bien qu’il partage une architecture structuralement proche des CRL (Cullin-RING), l’APC/C n’est pas une CRL au sens strict, car il n’utilise aucune culline canonique et ne dépend pas du cycle CSN-CAND1-NEDD8 (
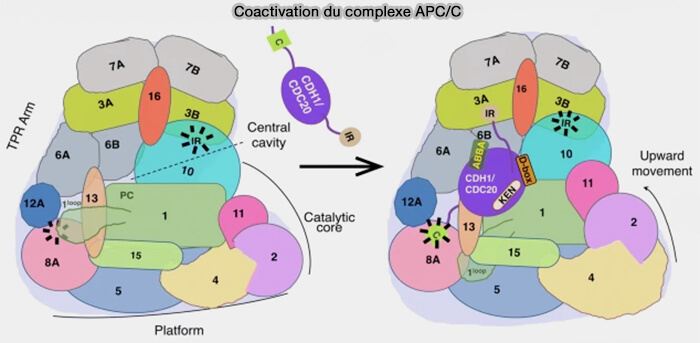
(Figure : vetopsy.fr d'après Bansal et Tiwari)
Module de reconnaissance (APC10 et coactivateurs)
Le module de reconnaissance relie la plateforme TPR au site catalytique.
1. Le module de reconnaissance comprend :
- APC10 (Doc1),
- les coactivateurs Cdc20 et Cdh1, qui ne font pas partie du complexe et dont le domaine W40 est responsable de la reconnaissance des degrons, i.e. D-Box, KEN-box et motif ABBA.
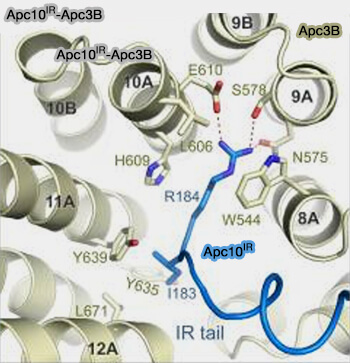
(Figure : vetopsy.fr d'après Cronin et coll)
2. APC10 et les coactivateurs Cdc20/Cdh1 forment ensemble la " poignée de reconnaissance du substrat ", i.e. coactivator-coreceptor module (Atomic structure of the APC/C and its mechanism of protein ubiquitination 2016).
a. Le corps globulaire d’APC10 établit des contacts hydrophobes et ioniques avec la face interne d’APC3 et la région adjacente d’APC1.
- Ces liaisons ancrent fermement APC10 à la base du module TPR, dans la zone médiane du complexe.
- Ce positionnement place APC10 à proximité immédiate du coactivateur (Cdc20 ou Cdh1), formant le module de co-reconnaissance du substrat.
b. L'extrémité C-terminale d'APC10 contient une queue IR (Isoleucine-Arginine) qui s’insère dans le même type de poche hydrophobe utilisée par les queues IR des coactivateurs (Cdc20/Cdh1).
- Cette poche IR se trouve sur la seconde copie d’APC3 (APC3B), formant un site de liaison symétrique avec celui utilisé par les coactivateurs sur APC3A.
- Cette interaction stabilise la plateforme TPR et aligne spatialement APC10 et le domaine WD40 du coactivateur au sein du module de co-reconnaissance du substrat.
Module catalytique (APC2, 11)
Le module catalytique, appelé soucent CRL (cullin-RING ligase) comprend :
- APC2, une sous-unité culline-like apparentée aux cullines des CRL (famille des ligases Cullin-RING),
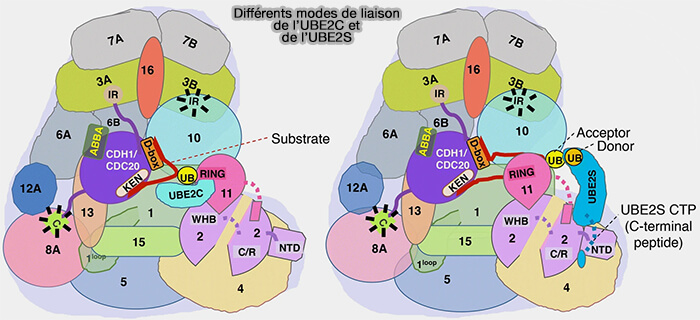
- APC11, une sous-unité RING qui assure le recrutement de l’E2 conjuguée à l’ubiquitine (Ube2C ou Ube2S).
Ce module organise le site catalytique et en assure la flexibilité structurale, condition essentielle pour le transfert d’ubiquitine sur les substrats reconnus.
APC2
Structure d'APC2
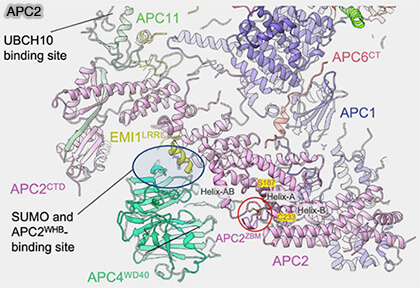
(Figure : vetopsy.fr d'après Höfler et Coll)
APC2 est une protéine culline-like de près de 900 acides aminés formée de trois domaines principaux.
1. Le domaine N-terminal hélicoïdal (NTD) est formé de plusieurs hélices α disposées en faisceau, servant de charpente d’ancrage aux sous-unités périphériques du complexe, en particulier APC4 et APC5.
Lors des changements conformationnels, ce domaine demeure relativement stable.
2. Le domaine central (CCD) est une région en hélice allongée, analogue au cullin repeat des CRL, responsable du maintien de la distance entre les modules de reconnaissance des substrats et le module RING.
a. Ce bras structural, plus mobile, est connecté au domaine WHB situé en C-terminal.
b. À ce niveau, APC2 établit un contact fonctionnel avec le module APC4-APC5, formant un nœud structural stable qui relie la plateforme TPR au module catalytique et cette interface APC2-APC4/5 joue un rôle mécanique clé.
- Elle transmet les mouvements des coactivateurs (Cdc20 ou Cdh1) vers le bras APC2-APC11,
- Elle permet la rotation du domaine WHB, orientant le RING d’APC11 vers le site E2~Ub,
- Elle stabilise la transition conformationnelle du complexe de l’état fermé (CRL-down) à l’état actif (CRL-up).
3. Le domaine C-terminal à hélice-ailée de type B (WHB) d’APC2, relié de manière flexible au corps de la protéine via le domaine C-terminal (APC2CTD), établit un contact direct avec le domaine RING d’APC11, formant ainsi le centre catalytique du complexe.
Remarque : APC2 contient un ion Zn++ coordonné par 4 cystéines (APC2ZBM) qui joue un rôle structural de stabilisation, comparable à celui observé dans certaines cullines classiques comme culline1 (CUL1) ou CUL3 (Cryo-EM structures of apo-APC/C and APC/CCDH1:EMI1 complexes provide insights into APC/C regulation 2024).
- Il contribue au positionnement correct du domaine WHB, garantissant une orientation stable du RING d’APC11 lors de la catalyse.
- Bien qu’il n’intervienne pas directement dans le transfert d’ubiquitine, il est indispensable à la cohésion du module APC2-APC11.
Organisation structurale d'APC2
1. L'organisation de l'APC2 en deux modules, NTD (ancrage) et CCD (bras structural), est essentielle pour :
- maintenir la distance correcte entre le centre de reconnaissance des substrats et le module catalytique,
- permettre le basculement du bras WHB-RING, pour orienter le site catalytique de l’APC/C de manière optimale pour le transfert de l’ubiquitine vers le substrat recruté.
- adapter la géométrie du site catalytique au type d’enzyme E2 engagé (Ube2C ou Ube2S).
2. La flexibilité intrinsèque du module catalytique constitue un élément clé du mécanisme enzymatique.
Elle permet d’ajuster la distance et l’angle entre APC11 et APC2, modulant la processivité de l’ubiquitination en fonction de l’état conformationnel du complexe et des partenaires associés, i.e. coactivateurs, E2 ou substrats.
- APC2CCD désigne la tige hélicoïdale (coiled-coil domain) formant la base du bras.
- APC2WHB (Winged Helix B) correspond à la charnière mobile pivotant autour du bras.
- APC11RING constitue le module catalytique terminal, responsable de la liaison E2~Ub.
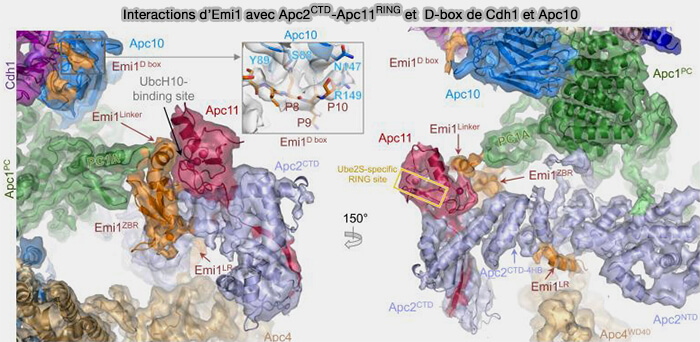
(Figure : vetopsy.fr d'après Chang et coll)
APC11
APC11 est une petite sous-unité d’environ 90 acides aminés, appartenant à la famille des ligases RING (Really Interesting New Gene).
Elle forme, avec APC2, le cœur catalytique du complexe APC/C, assurant le recrutement et l’activation des enzymes E2 chargées d’ubiquitine (Ube2C et Ube2S)
Structure d'APC11
1. APC11 adopte un repliement compact en double boucle RING, typique des E3 ligases de type RING. Sa structure est maintenue par un réseau de résidus cystéine et histidine qui coordonnent trois ions Zn²⁺.
a. Ces ions assurent :
- la stabilité tridimensionnelle du pli RING,
- la rigidité des boucles catalytiques RING1 et RING2,
- le positionnement optimal des résidus impliqués dans la liaison de l’E2 et le transfert d’ubiquitine en maintenant la topologie catalytique lors des transitions CRL-down → CRL-up.
b. Deux ions Zn²⁺ stabilisent les boucles catalytiques internes, tandis qu’un troisième participe à l’ancrage structural de la queue C-terminale d’APC11 sur le domaine WHB d’APC2, consolidant l’interface du module catalytique.
- L’absence de coordination zinc altère la géométrie du RING, abolit la liaison à l’E2 et rend le complexe catalytiquement inactif.
- Ainsi, les ions zinc d’APC11 sont directement catalytiques et structuraux, assurant la géométrie optimale du site de transfert d’ubiquitine.
2. Deux boucles forment une surface catalytique concave, où la cystéine active de l’E2~Ub est orientée face au substrat recruté par l’APC/C.
- La boucle N-terminale (RING1) sert de site de liaison primaire à l’enzyme E2, notamment Ube2C.
- La boucle C-terminale (RING2) assure le positionnement du bras catalytique par son interaction directe avec le domaine WHB d’APC2.
3. L’extrémité N-terminale d’APC11 interagit avec APC10 et, indirectement, avec les coactivateurs (Cdc20 ou Cdh1), renforçant le couplage entre reconnaissance du substrat et catalyse.
(Figure : vetopsy.fr d'après Yamaguchi et coll)
Organisation structurale d'APC11
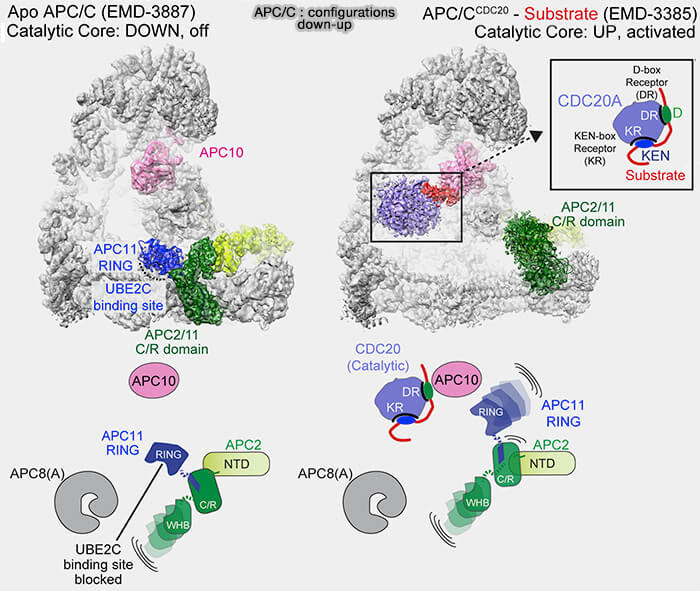
1. Le bord latéral d’APC11 interagit étroitement avec le domaine WHB d’APC2, créant un module catalytique flexible, capable de s’allonger ou de se replier selon l’état conformationnel du complexe (CRL-up ou CRL-down).
- Dans l’état apo (inactif), le RING d’APC11 est replié vers la plateforme TPR, désaligné du site de liaison E2~Ub.
- Lors de l’activation (CRL-up), l’extension du bras APC2-WHB repositionne le domaine RING d’APC11 face à l’E2, créant la géométrie catalytique nécessaire au transfert d’ubiquitine.
2. Cette flexibilité permet à l’APC/C d’adapter sa catalyse selon le type d’E2 recruté :
- Ube2C initie la mono-ubiquitination des substrats,
- Ube2S allonge les chaînes K11 pour la polyubiquitination processive.
(Figure : vetopsy.fr d'après Bansal et Tiwari)
Remarque : APC11 est structurellement apparenté aux petites sous-unités Rbx1 des ligases CRL classiques (CUL1-Rbx1, Cul3-Rbx1).
Cette homologie souligne la conservation évolutive du mécanisme de couplage cullin-RING, où le RING sert de plateforme universelle pour la catalyse du transfert d’ubiquitine.
États conformationnels du module catalytique
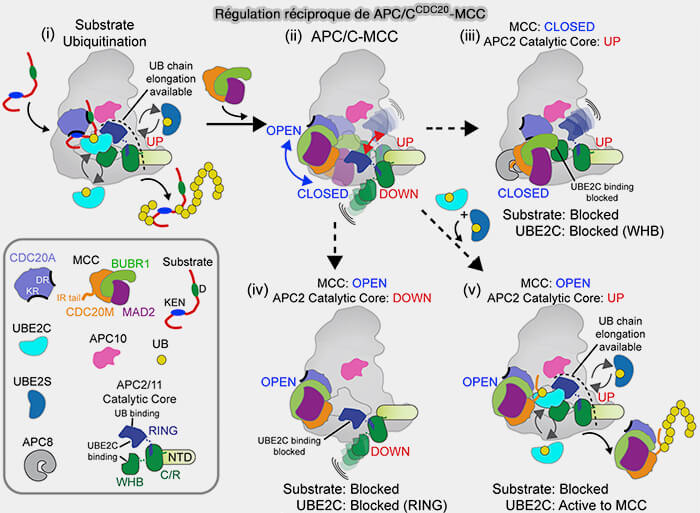
L’APC/C alterne entre trois états principaux :
1. La forme ouverte correspond à la conformation catalytiquement compétente. (A comparative study of the cryo-EM structures of S. cerevisiae and human anaphase-promoting complex/cyclosome (APC/C) 2024).
- Le bras APC2-APC11 est étendu.
- L’enzyme E2~Ub se positionne face au substrat.
- Les coactivateurs maintiennent la structure dans un état tendu et orienté, prêt à la catalyse.
(Figure : vetopsy.fr d'après Yamaguchi et coll)
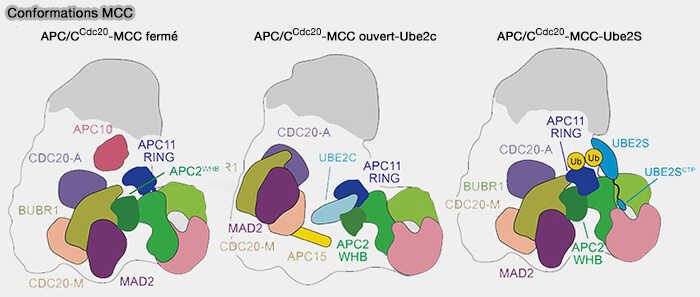
2. La forme relâchée (intermédiaire) est la phase de désengagement post-catalytique (Cryo-EM of Mitotic Checkpoint Complex-Bound APC/C Reveals Reciprocal and Conformational Regulation of Ubiquitin Ligation 2016).
- Le bras catalytique commence à se replier vers la plateforme TPR, tandis que certaines interactions avec les coactivateurs et les sous-unités du cœur persistent encore partiellement.
- Cette étape permet le relargage du substrat ubiquitiné et la libération progressive des E2.
(Figure : vetopsy.fr d'après Bodrug et coll)
3. La forme fermée est la conformation stable et verrouillée, i.e. forme apo sans coactivateur qui représente l’état basal de la ligase avant engagement (Cryo-EM structures of apo-APC/C and APC/CCDH1:EMI1 complexes provide insights into APC/C regulation (Cryo-EM structures of apo-APC/C and APC/CCDH1:EMI1 complexes provide insights into APC/C regulation 2024).
- Le module APC2-APC11 est complètement replié vers la plateforme TPR. et aucune interaction stable avec E2~Ub n’est maintenue.
- Le site catalytique est inaccessible.
| Terme structural |
Correspon- dances |
État fonctionnel | Bras catalytique (APC2-WHB + APC11-RING) |
|
|---|---|---|---|---|
| Bras relâché |
Fermé/ inactive apo-APC/C |
CRL-down/ inactive/ compact |
Inactif |
|
| Bras étendu |
Ouvert/ active |
CRL-up/ extended/ catalytique |
Actif |
|
| Bras intermédiaire |
Partiellement ouvert/ transition |
Intermediate/ pre-active |
Pré-activation |
|
Ces transitions conformationnelles du module catalytique, coordonnées par les coactivateurs et les enzymes E2, assurent le couplage entre la structure et la fonction du complexe APC/C, préparant le terrain pour le cycle catalytique décrit dans le mécanisme d’action.
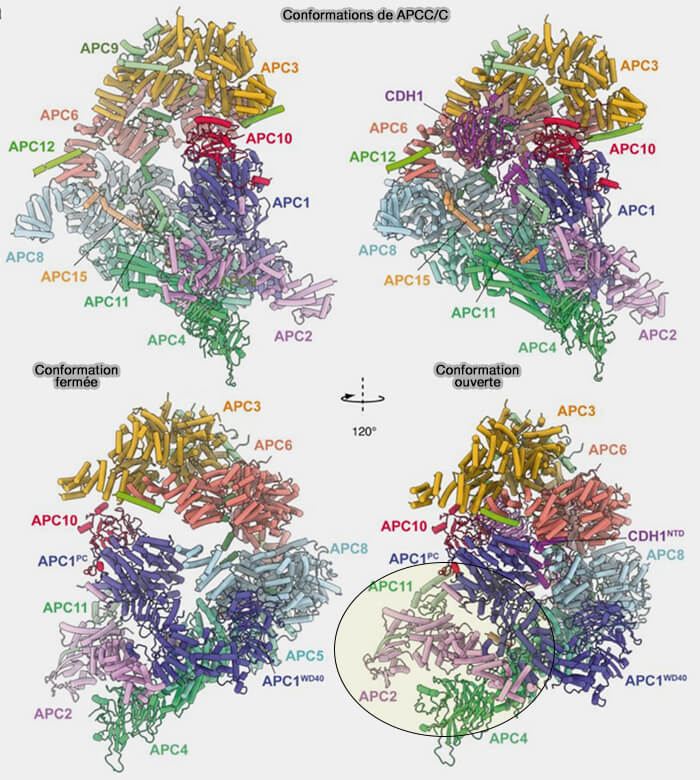
(Figure : vetopsy.fr d'après Vasquez-Fernadez et coll)
Conclusion structurale
1. L’organisation tridimensionnelle de l’APC/C définit une cavité centrale bordée par la plateforme et l’échafaudage TPR, au sein de laquelle s’articulent les modules de reconnaissance et catalytique.
- Le coactivateur et APC10 occupent la partie supérieure de cette cavité, en interaction étroite avec APC1, formant le module de co-reconnaissance du substrat.
- Le noyau catalytique APC2-APC11 se situe en avant, orienté vers ce module, dans une configuration favorable au transfert d’ubiquitine.
Cette architecture intégrée, combinant contacts multiples et flexibilité inter-domaines, confère à l’APC/C sa remarquable capacité d’adaptation conformationnelle. Elle permet la coordination dynamique entre reconnaissance, catalyse et régulation par les coactivateurs, les partenaires de liaison et les modifications post-traductionnelles.
2. Le tableau ci-dessous compare la composition du complexe APC/C chez les métazoaires et la levure.
Apc9 et Apc14 sont présentes chez la levure mais absentes chez l’Homme, tandis qu’Apc7 n’existe pas chez la levure.
- Chez les métazoaires, Apc9 est remplacé par APC4, APC5 et CDC26 qui stabilisent le grand arc du complexe.
- De même, Apc14 trouve son équivalent fonctionnel dans APC15 et APC16, impliquées dans la régulation dynamique du MCC et le détachement de Cdc20.
| Sous-unités de l'APC/C | Métazoaires | Levure (S. cerevisiae) |
|---|---|---|
| Module central | APC1 | Apc1 |
| APC4 | Apc4 | |
| APC5 | Apc5 | |
| APC15 | Apc15 | |
| Module TPR | APC3 | Cdc27 |
| APC6 | Cdc16 | |
| APC7 | - | |
| APC8 | Cdc23 | |
| APC12 | Apc12 | |
| APC13 | Apc13 | |
| APC16 | Apc16 | |
| CDC26 | Cdc26 | |
| Module reconnaissance | APC10 | Doc1 |
| Module catalytique | APC2 | Apc2 |
| APC11 | Apc11 | |
| Autres | - | Apc9 |
| - | Apc14 |



