Modifications post-traductionnelles des protéines
Ubiquitination : E3 ubiquitine ligases de la famille RING
Ligases Cullin-RING (CRL) : vue d'ensemble
- Biochimie
- Chimie organique
- Bioénergétique
- Composition de la matière vivante
- Composés organiques
- Protides
- Acides nucléiques
- Glucides
- Lipides
- Hormones
- Composés inorganiques
- Composés organiques
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

Les ligases CRL, E3 de la famille RING, construites autour d’une charpente en culline et d’un domaine RING catalytique, recrutent des adaptateurs spécifiques pour cibler les protéines à dégrader via le protéasome.
Vue d'ensemble des CRL
Les ligases Cullin-RING (CRL) forment la plus vaste sous-famille des E3 ubiquitine ligases de type RING, qui forment ainsi une plateforme dynamique de conjugaison de l’ubiquitine, intégrant la modularité structurale à la spécificité enzymatique.
1. Les CRL reposent sur une charpente modulaire en culline associée à :
- une sous-unité RING (Rbx1/Roc1) qui recrute l’enzyme de conjugaison E2,
- des adaptateurs variables (Skp1, Elongin, DDB1, BTB…) assurant la reconnaissance du substrat.
2. Cette organisation permet la formation d’une large diversité de complexes fonctionnels, parmi lesquels le complexe SCF (Skp1-Cullin1-F-box), pivot de la dégradation cyclique des régulateurs du cycle cellulaire.
Remarque : le complexe APC/C (Anaphase-Promoting Complex/Cyclosome), est une structure apparentée (CRL-like) qui n’est pas une CRL au sens strict, car il n’utilise aucune culline canonique et ne dépend pas du cycle CSN-CAND1-NEDD8 (
Son rôle est de controler de la progression mitotique.
Organisation modulaire des CRL
Les ligases Cullin-RING (CRL) partagent une architecture modulaire conservée articulée autour d’une protéine échafaudage de type culline, assurant la cohésion structurale et la coordination catalytique du complexe.
Charpente culline
Les cullines (CUL) constituent les protéines d’échafaudage des ligases CRL, assurant la stabilité structurale et la coordination catalytique du complexe.
Membres des cullines
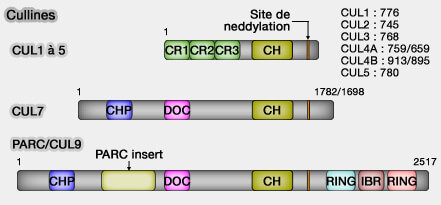
(Figure : vetopsy.fr d'après Sarikas)
La culline (CUL) est une protéine d'échafaudage (scaffold protein) qui comprend 8 membres chez les mammifères (The cullin protein family 2011) :
PARC, appelé aussi CUL9 contient un domaine RBR - RING/IBR/RING - et participe à quelque 200 E3.
Sur la figure ci-contre, les abréviations sont les suivantes : CH, Cullin Homology, CR, Cullin Repeat, CPH, domaine conservé dans CUL7, PARC et HERC2, DOC, domaine semblable à celui de l'APC.
| Culline | Complexe associé | Adaptateur principal | Facteur de spécificité du substrat | Rôle fonctionnel majeur |
|---|---|---|---|---|
| CUL1 | SCF (Skp1-Cullin1-F-box) | Skp1 | Protéines F-box | Dégradation cyclique (cycline E, p27Kip1) |
| CUL2 | VHL | Elongin B/C | Protéines à VHL-box | Réponse à l'hypoxie (HIF1α) |
| CUL3 | BTB | BTB | Protéines BTB-MATH | Homéostasie et signalisation cellulaire |
| CUL4A/B | DDB1-DCAF | DDB1 | Facteurs DCAF | Réparation de l'ADN, cycle cellulaire |
| CUL5 | SOCS | Elongin B/C | Protéines SOCS-box | Signalisation cytokines, immunité |
| CUL7 | Fbw8 | Skp1 | Protéines F-box spécifiques | Régulation de la croissance cellulaire |
| CUL9 (PARC) | - | - | - | Surveillance p53, apoptose |
Structure des cullines
CUL1, composée de 776 acides aminés, interagit avec différentes protéines (
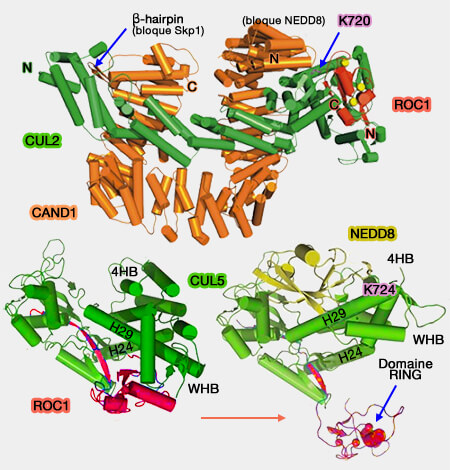
(Figure : vetopsy.fr d'après Goldenberg et Duda)
1. Son extrémité N-terminale, composée de trois répétitions Cullin Repeat (CR), recrute les adaptateurs de substrat (Skp1, Elongin B/C, DDB1, BTB…) qui assurent la sélectivité de reconnaissance.
- Chez CUL1, ce domaine interagit avec Skp1, qui connecte les protéines F-box du complexe SCF, modèle des CRL.
- Une organisation similaire est observée chez CUL7, où Skp1 recrute Fbw8, une protéine F-box atypique associée à la régulation du complexe mTORC2 (mTOR complex 2 regulates proper turnover of insulin receptor substrate-1 via the ubiquitin ligase subunit Fbw8 2012).
2. Son extrémité C-terminale contient un motif Cullin Homology (CH).
a. Le domaine WHB (Winged-Helix B), d'environ 200 résidus, est inclus dans ce domaine.
- Le domaine WHB comprend un repli UBL-like, qui contient la lysine conservée de neddylation, i.e. K720-K724 selon la culline.
- Il assure le positionnement du RING Rbx1/Roc1, indispensable au recrutement de l’E2 et au transfert d’ubiquitine.
b. Bien que le domaine WHB soit présent dans toutes les cullines, son rôle structurel est particulièrement marqué dans APC2, la culline du complexe APC/C.
Dans ce complexe, le WHB orchestre la bascule conformationnelle du module catalytique APC2-APC11, dont la fermeture/relaxation conditionne l’activation du complexe au cours de la mitose.
Remarque : certains membres, comme CUL7 ou PARC, comportent un domaine CPH (Conserved in PARC, HERC2 et CUL7) ou DOC (Domaine apparenté à l’APC), soulignant leur spécialisation fonctionnelle.
3. L’activité catalytique des cullines est finement régulée par des modifications post-traductionnelles, notamment la neddylation, ainsi que par des interactions modulatrices telles que celles de CAND1 (
Sous-unité RING (Rbx1/Roc1)
Les sous-unités Rbx1/ROC1 (Rbx1 - Ring box protein 1 - /Regulator Of Cullins 1) et Rbx2/ROC2 sont de petites protéines RING-finger d'une centaine de résidus qui constituent le noyau catalytique universel des ligases CRL.
- Elles assurent la liaison directe avec l’enzyme de conjugaison E2~Ub et positionnent le substrat pour le transfert de l’ubiquitine.
- Elles convertissent la plateforme structurale fournie par la culline en un centre enzymatique actif, où s’effectue la conjugaison finale de l’ubiquitine.
1. Rbx1/ROC1 est présente dans la majorité des complexes CRL, notamment CUL1 à CUL4.
- Son domaine RING coordonne deux ions Zn++ qui stabilisent la structure et forment la surface catalytique nécessaire à la fixation de l’E2 ( (ROC1, a Homolog of APC11, Represents a Family of Cullin Partners with an Associated Ubiquitin Ligase Activity 1999 et Structure of the cul1-rbx1-skp1-f boxskp2 scf ubiquitin ligase complex 2000).
- Elle agit comme un pont catalytique entre la culline (CUL) et l’enzyme de conjugaison E2~Ub, facilitant le transfert direct de l’ubiquitine sur la lysine du substrat.
- La flexibilité du lien entre Rbx1 et le domaine WHB de la culline permet un réajustement spatial du centre catalytique pendant le cycle de neddylation/déneddylation.
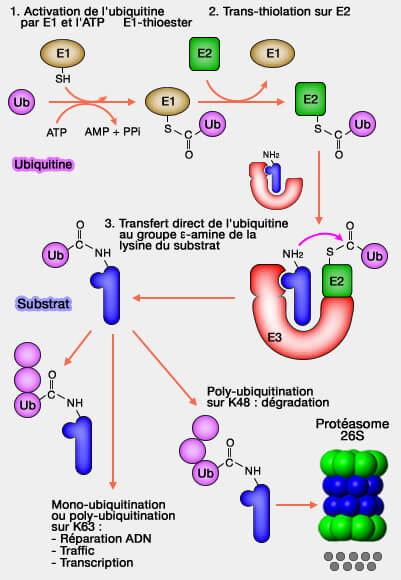
(Figure : vetopsy.fr)
2. Rbx2/Roc2 sont des paralogues fonctionnels de Rbx1, présents principalement dans les complexes CUL5 et certains CUL4-like.
Ils jouent un rôle équivalent dans le recrutement de l’E2 et la stimulation du transfert d’ubiquitine, mais avec des partenaires d’adaptation distincts, i.e. élongines B/C, SOCS-box… (ASB proteins interact with Cullin5 and Rbx2 to form E3 ubiquitin ligase complexes 2005).
Adaptateurs modulaires
Les ligases Cullin-RING (CRL) doivent leur grande polyvalence à un ensemble d’adaptateurs modulaires qui relient la charpente culline au module de reconnaissance du substrat.
Ces adaptateurs, souvent spécifiques d’un sous-type de culline, permettent de générer une vaste diversité de complexes E3 distincts tout en conservant une architecture catalytique commune.
Les principales famille d'adaptateurs :
- les adaptateurs Skps1 et les protéines F-box (complexe SCF / CUL1)
- les adaptateurs élongines B/C et SOCS-box (complexes ECS/CUL2 et CUL5)
- les adaptateurs BTB (complexe CUL3)
- les adaptateurs DDB1 et domaines DCAF/DWD Complexe CRL4 (CUL4A/B).

Tous ces adaptateurs sont étudiés dans un chapitre spécifique.
Régulation structurale et dynamique
L’activité des complexes Cullin-RING (CRL) est finement contrôlée par trois mécanismes complémentaires qui orchestrent l’état d’assemblage, la dynamique d’échange des récepteurs de substrats et la stimulation catalytique du complexe :
- la déneddylation par le complexe CSN,
- l’inhibition et le remodelage par CAND1,
- la réactivation par neddylation.
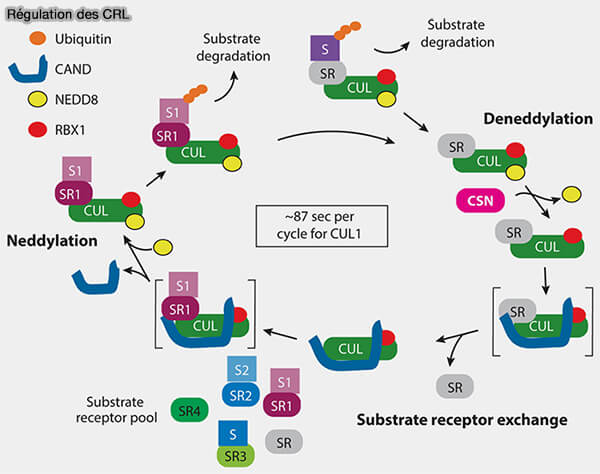
(Figure : vetopsy.fr d'après Shaaban et coll)
Déneddylation par le complexe CSN
1. Le complexe CSN (COP9 Signalosome) retire la protéine NEDD8 du domaine WHB de la culline, annulant la conformation active du module Cul-Rbx et plaçant le complexe dans un état inactif nécessaire à son réarrangement (Structural basis of Cullin 2 RING E3 ligase regulation by the COP9 signalosome 2018).
a. Le CSN est composé de huit sous-unités, i.e. CSN1-8, de masses moléculaires décroissantes de 57-22 kDa), et est organisé selon une architecture de main ouverte, qui présente une séquence et une homologie structurelle élevées avec le couvercle du protéasome (Structural Basis for a Reciprocal Regulation between SCF and CSN 2012 et Structural dynamics of the human COP9 signalosome revealed by cross-linking mass spectrometry and integrative modeling 2020).
- CSN1, 2, 3, 4, 7 et 8 sont structurellement homologues les uns aux autres et contribuent ensemble aux doigts de la main qui résultent de répétitions N-terminales α-hélicoïdales étendues.
- Chaque sous-unité CSN1-8 comprend une hélice C-terminale étendue qui s’associe pour former un faisceau hélicoïdal C-terminal.
- CSN5 et 6 sont également étroitement liés structurellement et forment un hétérodimère globulaire situé sur la paume de la main.
- Le CSN5 est responsable de l’activité déneddylase du CSN.
Remarque : une neuvième sous-unité, CNS9, appelée aussi CSNAP, a récemment été identifiée et jouerait un rôle dans la stabilisation du complexe CSN (CSNAP Is a Stoichiometric Subunit of the COP9 Signalosome 2015).
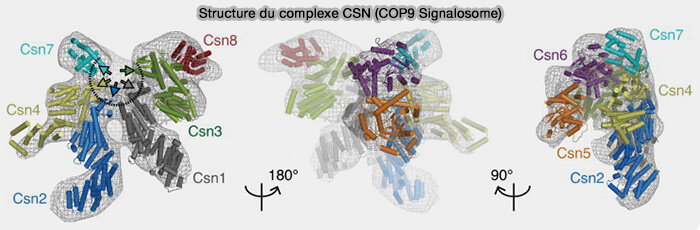
(Figure : vetopsy.fr d'après Enchev et coll)
a. La déneddylation de la culline :
- relâche la géométrie du module Cul-Rbx,
- rend la culline accessible à CAND1,
- autorise l’échange ou le relâchement des récepteurs de substrats, F-box, DCAF, BTB…,
- interrompt temporairement l’activité ubiquitine-ligase.
b. La déneddylation par le CSN constitue ainsi le point d’entrée du cycle dynamique des CR, préalable indispensable à l’intervention de CAND1 et au renouvellement des récepteurs (Structural and kinetic analysis of the COP9-Signalosome activation and the cullin-RING ubiquitin ligase deneddylation cycle 2016).
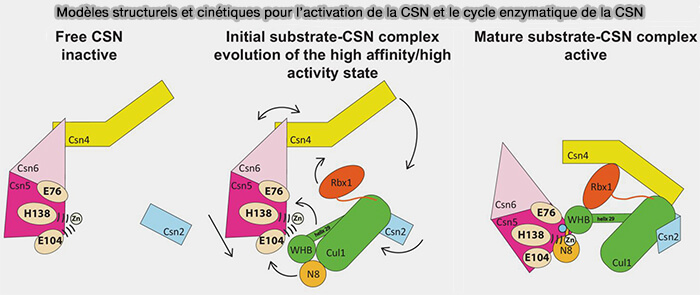
(Figure : vetopsy.fr d'après Mosadeghi et coll)
Inhibition et le remodelage par CAND1
Le régulateur CAND1 (Cullin-associated and Neddylation-Dissociated 1) se lie de manière sélective aux cullines non neddylées (CUL1, CUL2, CUL3, CUL4A et CUL5) pour inhiber leur assemblage (Molecular mechanisms of CAND2 in regulating SCF ubiquitin ligases 2025).
1. Il s’enroule autour de la culline et empêche la formation du complexe actif en masquant simultanément :
- le site de liaison à l’adaptateur, i.e. Skp1, DDB1, élongines…,
- la zone d’interaction avec les récepteurs de substrats, i.e. F-box, DCAF, BTB…,
- le site de neddylation porté par la lysine conservée du domaine WHB (Winged-Helix B) de la culline.
Cette conformation :
- empêche l’assemblage catalytique avec Rbx1 et l’E2~Ub,
- bloque toute activité ubiquitine-ligase,
- prépare l’échange du récepteur de substrat en créant un état permissif au remplacement des modules F-box, DCAF ou BTB.
2. Cette étape neutralise l’activité catalytique du complexe et prépare l’échange du récepteur de substrat.
Réactivation par neddylation
L’activation des CRL repose sur la neddylation, c’est-à-dire la conjugaison covalente de la petite protéine NEDD8 sur la lysine conservée du domaine WHB (Winged-Helix B) des cullines (Protein neddylation and its role in health and diseases 2024).
Ce domaine WHB adopte un repli UBL (Ubiquitine Like Protein), caractérisé par une topologie β-GRASP foldsimilaire à celle de l’ubiquitine, bien que sa séquence primaire diffère (Structural Insights into NEDD8 Activation of Cullin-RING Ligases: Conformational Control of Conjugation 2008 et Structure of a RING E3 Trapped in Action Reveals Ligation Mechanism for the Ubiquitin-like Protein NEDD8 2014 et Protein neddylation: beyond cullin-RING ligases 2015).
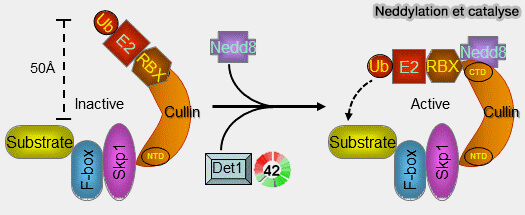
(Figure : vetopsy.fr d'après Malaria Parasite Metabolic Pathways)
1. Cette neddylation s'effectue sur la lysine conservée K724 du domaine WHB de CUL1.
2. Cette modification induit une réorganisation conformationnelle du complexe CUL-Rbx, provoquant :
- la libération du verrou CAND1,
- la bascule du domaine RING (Rbx1/Roc1) vers une position catalytique rapprochée,
- l’alignement optimal de l’E2~Ub face au substrat,
- la réactivation du complexe pour initier et allonger les chaînes d’ubiquitine.

La neddylation agit comme un interrupteur moléculaire UBL-like, permettant le passage du CRL d’un état inactif verrouillé, i.e. CUL déneddylée-CAND1-, à un état actif catalytique, i.e. CUL NEDD8.


