Cofacteurs
Coenzymes de transfert
Acétyl-CoA : biosynthèse à partir du pyruvate
Complexe pyruvate déshydrogénase (PDH) : E2 et E3
- Biochimie
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

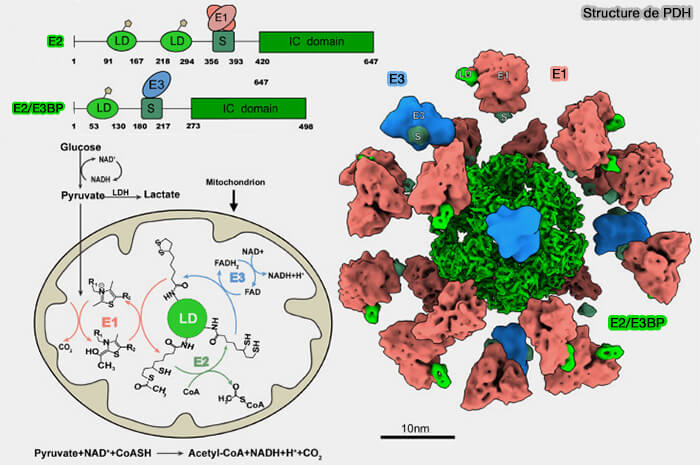
Le complexe pyruvate déshydrogénase (PDH), après l'action de E1 qui décarboxyle le pyruvate, transfère le groupe hydroxyéthyle à la lipoamide liée à E2, qui le transmet au coenzyme A (CoA) pour former l'acétyl-CoA , le dihydrolipoamide réduit étant ensuite régénéré en lipoamide par E3.
| Enzyme | Abrev. | Cofacteurs |
|---|---|---|
| Pyruvate déshydrogénase (PDH) (EC 1.2.4.1 : décarboxylase) |
E1 | Thiamine pyrophosphate (TPP) |
| Dihydrolipoyl/dihyrolipoamid S-acétyltransférase (DLAT) (EC 2.3.1.12 : acyltransférase) |
E2 |
|
| Dihydrolipoyl/dihyrolipoamide déshydrogénase (DLD) (EC 1.8.1.4 : oxydo-réductase) |
E3 |
Dihydrolipoyl/dihydrolipoamide S-acétyltransférase (DLAT ou E2)
Réaction
1. La dihydrolipoyl/dihydrolipoamide S-acétyltransférase (DLAT) ou dihydrolipoyl transacétylase (E2), EC 2.3.1.12, est une acyltransférase qui permet le transfert du groupe acétyle ($\ce{-C(=O)-CH3}$) sur la coenzyme A. Elle fait intervenir trois cofacteurs :
- la thiamine pyrophosphate (TPP), liée à E1,
- le lipoamide,
- la coenzyme A (CoA-SH).
$\ce{Acétyle-S-dihydrolipoamide + CoA_SH}$ $\longrightarrow$ $\ce{Acétyl−CoA + Dihydrolipoamide}$
2. La liaison thioester " riche en énergie " permet :
- au groupe acétyle d'être transféré du groupe thiol du dihydrolipoamide, i.e. lipoamide réduit, sur le groupe thiol ou sulfhydryle ($\ce{R−SH}$) de la coenzyme A sous forme d'acétyl-CoA qui quitte le complexe enzymatique,
- d'obtenir du dihydrolipoamide réduit avec ses 2 ($\ce{R−SH}$) libres, qui reste attaché à E2 par la lysine,, qui sera régénéré en lipoamide par E3 (
structure du lipoamide).
Remarque : E2 a été aussi localisée dans le noyau cellulaire, indépendamment de PDH, et serait impliquée dans les voies de transduction STAT et pourrait influencer l’expression génique, la prolifération, la différenciation ou la survie cellulaire.
STAT est un transducteur de signal et un activateur de transcription qui jouent un rôle essentiel dans la réponse cellulaire à une grande variété de cytokines et de facteurs de croissance en régulant des gènes nucléaires spécifiques (Nuclear localization of pyruvate dehydrogenase complex-E2 (PDC-E2), a mitochondrial enzyme, and its role in signal transducer and activator of transcription 5 (STAT5)-dependent gene transcription 2012).
Structure de E2
(Figure : vetopsy.fr d'après Wang et coll)
Le complexe PDH comprend une quarantaine de E2, composées chacune de quatre domaines reliés par un lien flexible, décrits de l'extrémité N-terminale à l'extrémité C-terminale (Solution Structure and Characterisation of the Human Pyruvate Dehydrogenase Complex Core Assembly 2010).
Domaines lipoyl
Deux domaines lipoyl, le domaine lipoyle 1 (L1) et le domaine lipoyle 2 (L2) portent chacun un lipoamide lié de manière covalente à une lysine (
(Figure : vetopsy.fr d'après Wang et coll)
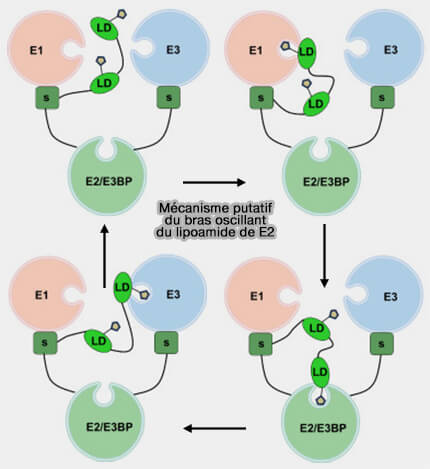
1. L1 est principalement impliqué dans le transfert du groupe acétyle entre les sites actifs des enzymes E1, E2 et E3, via un mécanisme de bras oscillant (swinging arm), caractéristique des enzymes multifonctionnelles (Swinging arms and swinging domains in multifunctional enzymes: catalytic machines for multistep reactions 2000).
2. L2, quant à lui, joue un rôle crucial dans la régulation de la phosphorylation de la sous-unité E1 par la PDK (
Domaines PSBD ou S
Le domaine de liaison sous-unitaire, PSBD (Peripheral Subunit Binding Domain), appelé aussi S, interagit spécifiquement avec les sous-unités E1 et E3, assurant leur ancrage autour du noyau E2.
1. Le PSBD d'E2 se lie à une poche conservée sur la sous-unité β de E1 par une hélice-épingle à cheveux-hélice (helix-turn-helix) qui s’encastre dans une rainure sur E1 (Binding of pyruvate dehydrogenase to the core of the human pyruvate dehydrogenase complex 2008).
Les liaisons sont de nature principalement hydrophobe, i.e. motif Leu-x-Leu souvent conservé sur E2, mais aussi ionique, Arg, Glu…, et hydrogène, assurant une affinité suffisante pour maintenir E1 attachée sans gêner la flexibilité du lipoyl.
2. Chez les eucaryotes, PSBD s’associe étroitement avec les protéines E3BP (E3-binding protein ou PDH Protein X), qui servent à ancrer la sous-unité E3 au complexe (Structural insight into interactions between dihydrolipoamide dehydrogenase (E3) and E3-binding protein of human pyruvate dehydrogenase complex 2010).
Elles s’intègrent dans le noyau central du complexe PDH en remplaçant certaines sous-unités E2, formant un assemblage stable.
a. Les E3BP partagent une architecture similaire avec la sous-unité E2, mais avec :
- avec un seul domaine lipoyle,
- un domaine catalytique qui ne possède pas d’activité catalytique acétyltransférase, i.e. les résidus de la triade Cys, Lys et His étant remplacés ou absents, ainsi que les AsP/Glu stabilisateurs remplacés par des résidus non chargés (Variation in the organization and subunit composition of the mammalian pyruvate dehydrogenase complex E2/E3BP core assembly 2011).
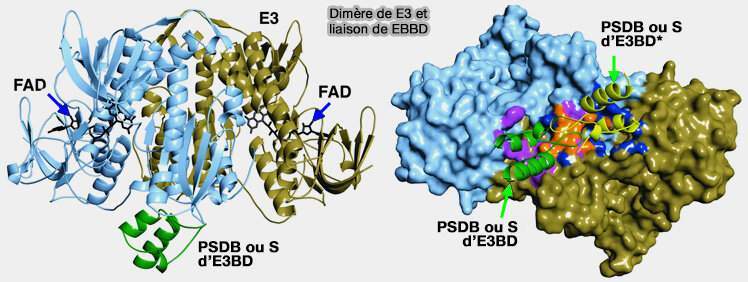
b. E3BP comporte un domaine de liaison spécifique qui s’insère dans une poche hydrophobe, au niveau de l'interface du dimère E3, i.e. un seul site de liaison par homodimère E3.
- L'interaction implique des résidus clés comme P133, P154 et I157 sur E3B, et les tyrosines Y438 sur E3.
- Les interactions périphériques sont R130–Y438/D444, S132–D444, N137–R447, K160–E443/D4', et E161–T412.
(Figure : vetopsy.fr d'après Brautigam et coll)
Domaine catalytique (IC)
Le domaine interne ou domaine catalytique (IC) est responsable de l'activité catalytique acétyltransférase et forme le coeur du complexe PDH.
Domaine de trimérisation
Le domaine de trimérisation à l'extrémité C-terminale permet la formation d'une structure dodécaédrique chez les eucaryotes, ou cubique chez les bactéries de 24 ou 60 sous-unités selon l’organisme.

Vous pouvez lire : Atomic Structure of the E2 Inner Core of Human Pyruvate Dehydrogenase Complex (2018).
Dans le cœur catalytique humain, la trimérisation implique une interaction " knob-and-hole ", i.e. bouton-pression hydrophobe, au niveau des hélices, entre :
- les extrémités C‑terminales des E2 d’un trimère, i.e. knob (Leu, Ile, Val, Phe) sur H7,
- des poches hydrophobes sur le trimère adjacent, i.e. hole (Ala, Tyr, Gly, Leu) formé par H2/H4.
Dihydrolipoyl/dihydrolipoamide déshydrogénase (DLD ou E3)
Réaction
La dihydrolipoyl/dihydrolipoamide déshydrogénase (DLD) (E3), EC 1.8.1.4, est une oxydo-réductase dont la coenzyme est la FAD (flavine adénine dinucléotide).
- Elle réoxyde le dihydrolipoamide de la transacétylase précédente en lipoamide.
$\ce{Dihydrolipoamide + NAD+}$ $\longrightarrow$ $\ce{Lipoamide + NADH + H+}$
Cette réaction s'effectue en deux étapes.
1. FAD réoxyde le dihydrolipoamide.
$\ce{Dihydrolipoamide + FAD}$ $\longrightarrow$ $\ce{Lipoamide + FADH2}$
- FAD, liée de manière covalente dans E3, accepte les deux électrons du dihydrolipoamide pour former FADH2.
- Le lipoate est réoxydé en lipoamide prêt pour un nouveau cycle catalytique.
2. FADH2 transfère les électrons à NAD+.
$\ce{FADH2 + NAD+}$ $\longrightarrow$ $\ce{FADH2 + NADH + H+}$
- La FADH2 sera réoxydée en FAD par le NAD+ qui se transforme en NADH.
- Une fois régénéré, le FAD peut participer au cycle de réaction suivant.
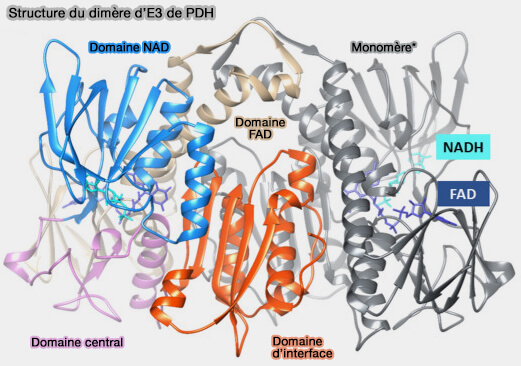
Structure de E3
E3 est un homodimère qui comprend 6-12 complexes (Dihydrolipoamide dehydrogenase, pyruvate oxidation, and acetylation-dependent mechanisms intersecting drug iatrogenesis 2021).
(Figure : vetopsy.fr d'après Duarte et coll)
1. Chaque sous-unité contient :
- un site FAD, lié de manière non covalente mais étroitement associé,
- un site NAD+ pour la régénération de FAD,
- un domaine central,
- une interface de liaison avec l'E3BP, i.e. la protéine E3BP se liant au niveau de l'interface du dimère E3, i.e. un seul site de liaison par homodimère E3 (
cf plus haut).
2. E3 remplit une fonction catalytique similaire dans plusieurs autres complexes qui emploient le lipoamide (
À chaque fois qu’un E2 utilise du lipoamide, il finit avec un dihydrolipoamide réduit, qui doit être réoxydé pour que le cycle catalytique continue.
a. Cependant, E3 se lie à la PDH beaucoup plus fortement grâce à un résidu asparagine, i.e. Asn137, qui stabilise l’architecture centrale du complexe (Structural insight into interactions between dihydrolipoamide dehydrogenase (E3) and E3-binding protein of human pyruvate dehydrogenase complex 2010).
b. Dans les autres complexes, il est remplacé par un résidu arginine, comme Arg114 dans la DLST humaine, beaucoup plus gros, qui perturbe l’ajustement optimal dans la poche de liaison de E3 :
- en réduisant l'affinité de liaison et la durée de l'interaction,
- en augmentant sa mobilité.
Cela permettrait d'adapter l'activité plus rapidemnent de ces complexes selon l’état métabolique.
Importance de la pyruvate déshydrogénase (PDH) et régulation
Biologie cellulaire et moléculaireConstituants de la celluleMatrice extracellulaireReproduction cellulaireBiochimieChimie organiqueBioénergétiqueProtidesAcides aminésProtéinesGlucidesLipidesEnzymesCofacteursCofacteurs d'oxydoréductionCoenzymesVitaminesHormonesTransport membranaireMoteurs moléculairesVoies de signalisation


