Enzymes
Groupe des hydrolases (EC 3)
ATPases
P-ATPases : mécanisme
- Biochimie
- Chimie organique
- Bioénergétique
- Composition de la matière vivante
- Composés organiques
- Protides
- Acides nucléiques
- Glucides
- Lipides
- Enzymes
- Vue d'ensemble
- Classification des enzymes
- Oxydoréductases (EC 1)
- Transférases (EC 2)
- Hydrolases (EC 3)
- Protéases (peptidases ou enzymes protéolytiques)
- Phosphatases
- Estérases
- Lipases
- Phospholipases
- ATPases
- Vue d'ensemble et classification des ATPases
- ATPases à moteur rotatif : F/V/A ATpases
- P-ATPases
- Vue d'ensemble et classification des P-ATPases
- P4-ATPases
- Protéines AAA et AAA+
- Transporteurs ABC
- E-ATPases
- GTPases
- Lyases ou synthases (EC4)
- Isomérases (EC5)
- Ligases ou synthétases (EC 6)
- Coenzymes
- Vitamines
- Hormones
- Composés inorganiques
- Composés organiques
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

Le mécanisme des P-ATPases diffèrent des autres ATPases par la présence d'un aspartate conservé qui subit une phosphorylation transitoire au cours du cycle ATP, d'où le nomde type type P.
Vue d'ensemble du mécanisme des P-ATPases

Vous pouvez lire : P-type ATPases use a domain-association mechanism to couple ATP hydrolysis to conformational change (2019), article dans lequel le mécanisme complexe est détaillé. Nous en ferons un résumé succinct.
1. Les ATPases de type P fonctionnent selon un modèle « à accès alterné » (
- Dans ce modèle, les voies d'accès aux deux côtés de la membrane sont transitoirement fermées pour enfermer les ions avant de s'ouvrir de chaque côté de manière alternée.
- La translocation ionique réelle est accomplie par des changements conformationnels importants provoqués par l'hydrolyse de l'ATP.
L'ATPase alterne généralement entre deux conformations, les états dits E1 et E2.
- Les états E1 sont associés à l'autophosphorylation par l'ATP et ont une forte affinité pour le cation qui doit être expulsé du cytoplasme.
- Les états E2 sont associés à l'autodéphosphorylation, ont une plus faible affinité pour ces cations et peuvent se lier à des contre-ions à la place.
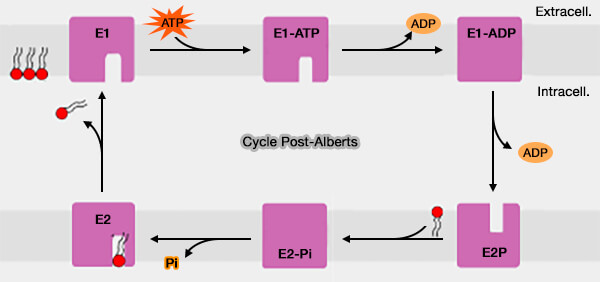
(Figure : vetopsy.fr d'après Cheng et coll)
2. Conceptuellement, les phosphorylations/déphosphorylations ressemblent aux événements de protonation et de déprotonation dans les transporteurs secondaires entraînés par la force motrice du proton ou PMF (Thermodynamic secrets of multidrug resistance: A new take on transport mechanisms of secondary active antiporters 2018).
En d'autres termes, la P-ATPase continuerait à parcourir le chemin de E1 → E1P → E2P → E2 → E1 indépendamment du fait que la liaison au substrat se soit produite, tant que l'hydrolyse de l'ATP peut être découplée du transport du substrat.
Mécanisme proprement dit
Les interactions entre les trois domaines cytosoliques changent en réponse au chargement et à la libération du substrat.
1. Le résidu aspartate clef dans un motif DKTGT conservé du domaine P (par exemple, Asp351 dans SERCA1, alterne entre les états phosphorylé et déphosphorylé.
a. Le domaine N fonctionne comme une kinase intégrée (enzyme-1 ou E1) pour transférer le groupe γ-phosphate de l'ATP au domaine P.
Cette réaction est catalysée en utilisant un mécanisme SN2 médié par deux cofacteurs Mg++ liés au domaine P (site I, stabilisant le γ-phosphate ainsi que le résidu clef Asp) et au domaine N (site II, liaison avec le α- et β-phosphates d'ATP ou d'ADP).
b. en utilisant son motif TGES conservé, le domaine A fonctionne comme une cis-phosphatase pour catalyser la déphosphorylation. Cette dernière réaction utilise également un mécanisme SN2 mais est facilitée par un seul cofacteur Mg++ lié au site I (How processing of aspartylphosphate is coupled to lumenal gating of the ion pathway in the calcium pump 2007).
Comme les modules kinase et phosphatase ciblent le même résidu Asp dans le domaine P, les domaines N et A doivent commuter activement avec le domaine P, pour se concerter sur le prochain événement déclencheur, i.e. chargement ou libération du substrat pour soutenir les cycles d'hydrolyse de l'ATP.
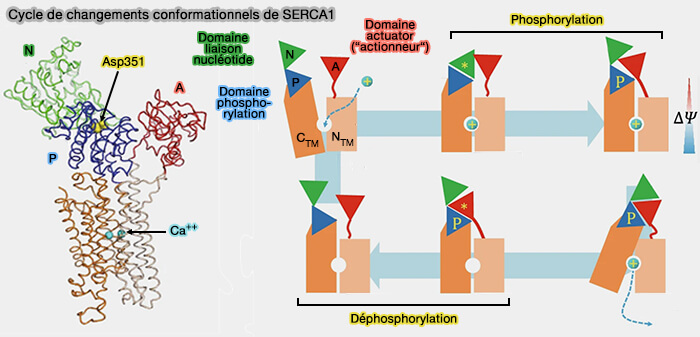
(Figure : vetopsy.fr d'après Zhang et coll)
2. Dans un certain nombre de structures P-ATPase, une hélice amphipathique (AH) est observée.
a. À l'extrémité N-terminale de M1, elle est susceptible d'ancrer le sous-domaine NTM à la bicouche lipidique, limitant particulièrement le mouvement de l'extrémité N-terminale de M1 à la surface du feuillet cytosolique (Crystal structure of the high-affinity Na+,K+-ATPase– ouabain complex with Mg 2+ bound in the cation binding site 2013),.
b. À l'extrémité luminale de M9 ou M10 dans les P4-ATPases, on la trouve dans plusieurs grandes familles de protéines membranaires comme :
- les transporteurs ABC (Thermodynamic secrets of multidrug resistance: A new take on transport mechanisms of secondary active antiporters 2018),
- les récepteurs couplés aux protéines G ou GPCR (Proton transfer-mediated GPCR activation 2014),
- les canaux ioniques voltage-dépendants (Thermodynamics of voltage-gated ion channels 2018).
Ces hélices amphipathiques sont susceptibles de jouer un rôle important en limitant le mode de changements conformationnels de leurs extrémités d'hélice TM connectées au glissement uniquement sur la surface de la membrane (Interplay between the electrostatic membrane potential and conformational changes in membrane proteins 2019).
Remarque : en outre, les interactions protéine-phospholipide sont régies aussi par des tryptophanes ainsi que par des résidus basiques qui exercent leur effet stabilisateur sur les protéines membranaires par le biais d'interactions entre les dipôles électriques intrinsèques (et induits) de leurs chaînes latérales indole et le fort champ électrique local des phosphates du groupe de tête des molécules lipidiques environnantes.
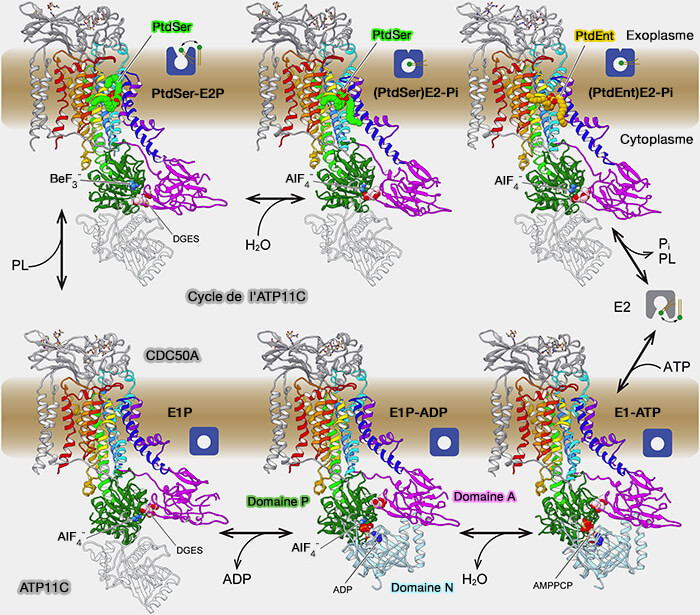
(Figure : vetopsy.fr d'après Nakanishi et coll)
3. Dans un cycle fonctionnel de la P-ATPase, la liaison et la libération du substrat sont considérées comme des conditions nécessaires pour les événements de phosphorylation et de déphosphorylation, respectivement.
a. Cependant, les deux types d'événements se produisent à 45 Å de distance l'un de l'autre.
Ainsi, une question centrale dans le domaine de recherche sur la P-ATPase concerne le(s) mécanisme(s) de communication entre les sites de liaison au substrat et de phosphorylation (Conformational transitions and alternating-access mechanism in the sarcoplasmic reticulum calcium pump 2017 et Thermodynamics of voltage-gated ion channels 2018).
b. Les études de biologie structurale ont fourni des informations essentielles pour répondre à cette question.
Par exemple, SERCA1 contient deux sites de liaison Ca++ (Crystal structure of the calcium pump of sarcoplasmic reticulum at 2.6 A resolution 2000).


