Domaines protéiques liés à l'ubiquitine
UBR-Box et N-domaine

Les UBR-box et les N-domaines sont inclus dans les N-recognines qui reconnaissent les N-degrons de l'Arg/règle N-terminale.
La voie de la règle du N-terminal (N-end rule) est un système protéolytique : la déstabilisation de certains résidus N-terminaux peut donner un signal pour la destruction de la protéine par le protéasome 26S : elle nécessite une ubiquitination chez les eucaryotes.
N-recognines (N-recognin)
1. Les N-recognines sont des protéines qui reconnaissent les N-degrons de l'Arg/règle N-terminale (Arg/N-end rule) qui possèdent (The Substrate Recognition Domains of the N-end Rule Pathway 2009 et The N-end rule pathway: From recognition by N-recognins, to destruction by AAA + proteases 2012) :
- des UBR-box qui reconnaissent les N-degrons de type 1 (arginine, lysine et histidine : la cystéine, l'asparagine et la glutamine sont transformées),
-
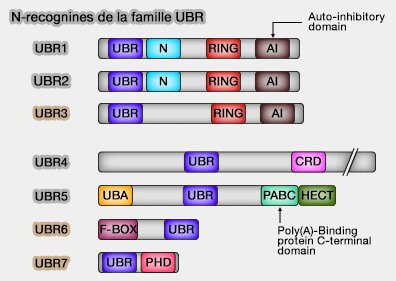
Protéines UBR
(Figure : vetopsy.fr)
L'Ac/règle N-terminale (Ac/N-end rule), autre voie de la règle du N-terminal, est déjà présente chez les Procaryotes, ainsi que la leu/N-end rule chez les bactéries.
2. Les protéines UBR sont des E3 ubiquitine ligases qui comptent 7 membres chez les mammifères (1 seul chez la levure).
a. UBR 1/2/4/5 sont des N-recognines (A Family of Mammalian E3 Ubiquitin Ligases That Contain the UBR Box Motif and Recognize N-Degrons 2005).
Ubr5 ne reconnait que le type 1 alors que les autres reconnaissent les deux types.
b. UBR3, 6 et 7 n'en sont pas, bien qu'elles possèdent aussi une UBR-box (The Substrate Recognition Domains of the N-end Rule Pathway 2009).
UBR-Box
1. la structure de l'UBR-Box, d'environ 70 à 80 acides aminés, est composée (Structural basis for the recognition of N-end rule substrates by the UBR box of ubiquitin ligases 2010 et Structural basis of substrate recognition and specificity in the N-end rule pathway 2010 et The UBR-box and its relationship to binuclear RING-like treble clef zinc fingers 2015) :
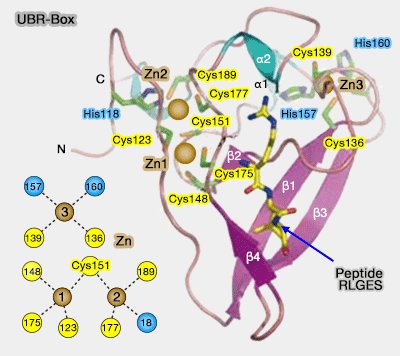
(Figure : vetopsy.fr d'après Choi)
a. d'un coeur compact avec trois sites de chélation du zinc
b. d'une forme en V, délimitée par deux brins β (β strand) antiparallèles et deux boucles irrégulières.
Les UBR humaines contiennent 2 hélices α, et les levures 3 hélices 310.
c. La structure du domaine U-box est proche du domaine RING (
2. Les UBR se lient et reconnaissent les N-degrons de type 1, i.e. les arginine, lysine ou histidine N-terminales.
- Tous les résidus de la UBR-box semblent nécessaires et suffisants pour la liaison (The N-End Rule Pathway 2012).
- Par contre, la mutation de Asp150 semble abolir complètement la liaison au substrat (ici, peptide RLGES).
N-domaine
Le N-domaine est une région adjacente à l'UBR-box des N-recognines qui reconnaît les N-degrons de type 2 (leucine, isoleucine, phénylalanine, tryptophane et tyrosine).
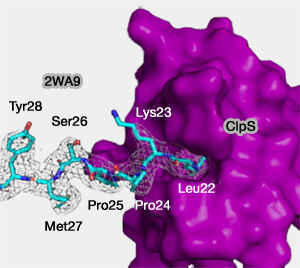
(Figure : vetopsy.fr d'après Roman)
1. Ce N-domaine d'environ 80 résidus présente une similitude structurelle et fonctionnelle avec la N-recognine d'Escherichia coli, ClpS (appelée aussi ATP-dependent Clp protease adaptor protein).
Chez la levure, cette Clps se lie au N-degron et présente la protéine à la protéase ClpAP ATP-dépendante (The ClpS Adaptor Mediates Staged Delivery of N-End Rule Substrates to the AAA+ ClpAP Protease 2011).
2. Par contre, contrairement à l'UBR-box, le N-domaine seul ne suffit pas à la liaison au substrat, ce qui laisse supposer que certains résidus spécifiques de ce domaine doivent être proches de la structure tridimensionnelle de l'UBR-box.
Retour aux domaines protéiques
Biologie cellulaire et moléculaireConstituants de la celluleMatrice extracellulaireReproduction cellulaireBiochimieChimie organiqueBioénergétiqueProtidesAcides aminésProtéinesDomaines protéiquesModifications post-traductionnelles des protéinesDégradations des protéinesUbiquitinationGlucidesLipidesEnzymesCoenzymesVitaminesHormonesTransport membranaireMoteurs moléculairesVoies de signalisation


