Constituants cellulaires : protéasome 26S
Structure du complexe régulateur 19S
- Biologie cellulaire et moléculaire
- Constituants de la cellule
- Membrane plasmique
- Noyau
- Cytoplasme
- Hyaloplasme
- Cytosol
- Cytosquelette
- Proteasome
- Vue d'ensemble
- Structure générale du protéasome 26S
- Assemblage du protéasome 26S
- Fonctionnement du protéasome
- Morphoplasme : organites
- Hyaloplasme
- Matrice extracellulaire
- Reproduction cellulaire
- Biochimie
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

Le pore de l'entrée du coeur catalytique (CP : Core Particle) du protéasome est fermé, un peu comme le fait un " couvercle " sur une marmite, par des complexes régulateurs (RP : Regulatory Particle) dont le principal est le complexe 19S.
L'acceptation du substrat polyubiquitiné active la RP pour l'ouvrir.
Le protéasome 26S est un protéasome comprenant un CP 20S et 1 ou 2 RP 19S - PA700 - (Recognition and Processing of Ubiquitin-Protein Conjugates by the Proteasome 2009). D'autres complexes régulateurs existent comme PA200/Blm10 ou le complexe 11S (REG ou PA28).
Le complexe 19S (PA700) est une structure hautement conservée, présente dans les cellules eucaryotes.
- Il reconnaît les ubiquitines de la protéine polyubiquitinée (cf. acceptation du substrat),
- Il hydrolyse l'ATP pour déplier et transloquer les protéines vers le coeur catalytique (cf. engagement et translocation du substrat),
- Il clive l'ubiquitine (désubiquitination) qui peut être alors recyclée (cf. désubiquitination du substrat).
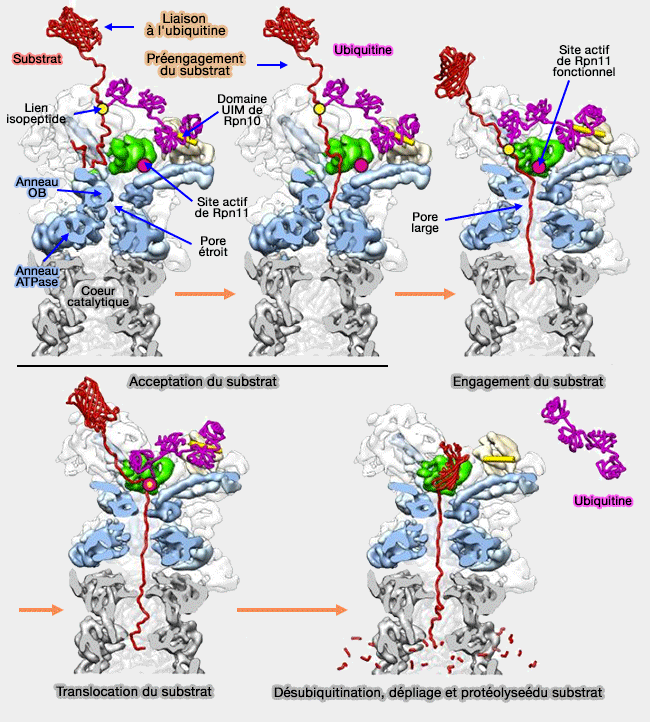
(Figure : vetopsy.fr d'après Lander)
Vue d'ensemble du complexe 19S
Le complexe 19S (RP : Regulatory Particle) est constituée d'au moins 19 protéines distinctes réparties dans deux unités.
1. La base contient 10 protéines :
- 6 Rpt : 1 à 6 (Regulatory particle triple-A protein ou Regulatory particle triphosphatase)
- 4 Rpn : 1, 2, 10, 13 (Regulatory particle non-ATPase).
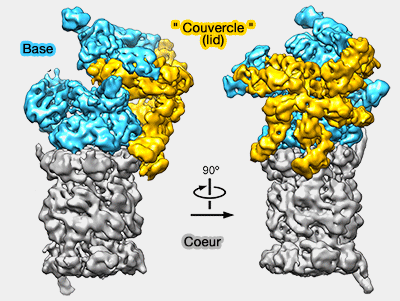
(Figure : vetopsy.fr d'après Lander)
2. Le " couvercle " (lid) en comporte 8 (ou 9)
- 6 à domaine PCI : Rpn3, 5, 6, 7, 9, 12
- 2 à domaine MPN : Rpn8 et 11,
- Rpn15 (Sem1).
La nomenclature a été unifiée (Unified nomenclature for subunits of the Saccharomyces cerevisiae proteasome regulatory particle 1998).
Le protéasome est une structure dynamique qui modifie sa conformation durant le processus et qui interagit avec un grand nombre de protéines.
- La description de la base et du " couvercle " (lid) concerne surtout la nomenclature, la structure et la conformation des différentes protéines.
- Leur emplacement les unes par rapport aux autres sont encore en cours d'étude. Toutefois, les positions décrites sont celles supposées du protéasome libre de tout substrat.
Les changements conformationnels ou positionnels sont décrits dans le fonctionnement du protéasome.
Base du complexe 19S
Biologie cellulaire et moléculaireMembrane plasmiqueNoyauCytoplasmeRéticulum endoplasmiqueAppareil de GolgiMitochondriesEndosomesLysosomesPeroxysomesProtéasomesCytosqueletteMicrofilaments d'actineFilaments intermédiairesMicrotubulesMatrice extracellulaireReproduction cellulaireBiochimieTransport membranaire Moteurs moléculairesVoies de signalisation


